データガバナンス機能では、以下のような操作で研究・実験を進めていきます。
本機能が想定する研究・実験については『想定する研究・実験について』を参照ください。
- ユーザー登録を行う
- 研究を開始する(①、②、③)
- 実験を開始する(④、⑤、⑥、⑦)
- 実験を途中保存する(⑧)
- 実験の説明を記述する
- 実験を再現するためにSnakefile(タスクランナー)を記述する
- 実験を終了する
- メタデータを検証する(4~7と並行実施可)
- 3~7を繰り返して研究を進める
- 研究を終了する
- 複数の研究を実施したい場合は2~9を繰り返す(並行実施可)
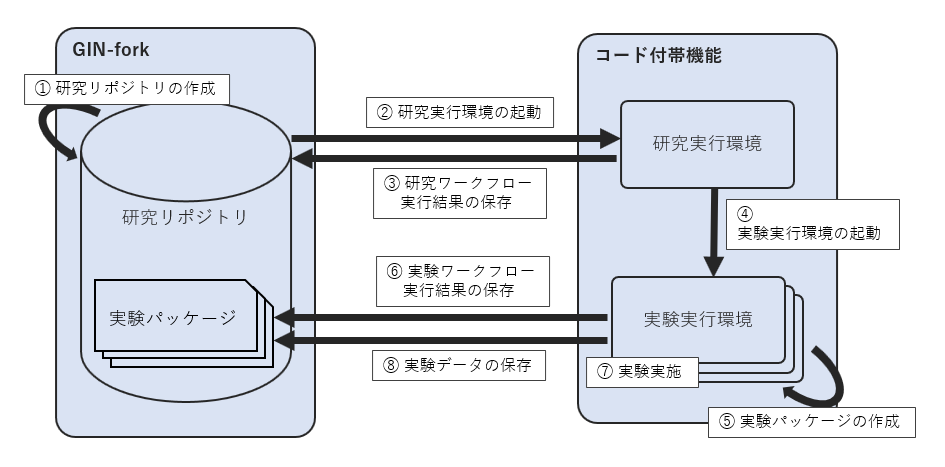
付録1 GIN-forkの操作について
付録2 Jupyter Notebookについて
付録3 その他機能について
1. ユーザー登録を行う
データガバナンス機能を利用するには、データ解析機能を利用できるアカウントが必要です。
データ解析機能を利用できるアカウントをご用意の上、以下の手順に従って利用を開始してください。
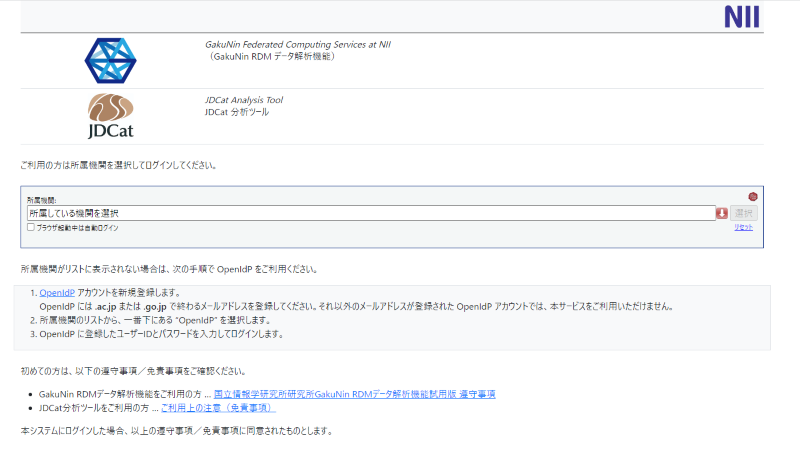
- GIN-forkのページへアクセス
https://dg.nii.ac.jp/ へブラウザでアクセスしてください。
- ユーザー登録画面へ遷移
ユーザー登録を行います。アクセスした先のページ右上にある「登録」ボタンを押下して、ユーザ登録画面へ遷移してください。
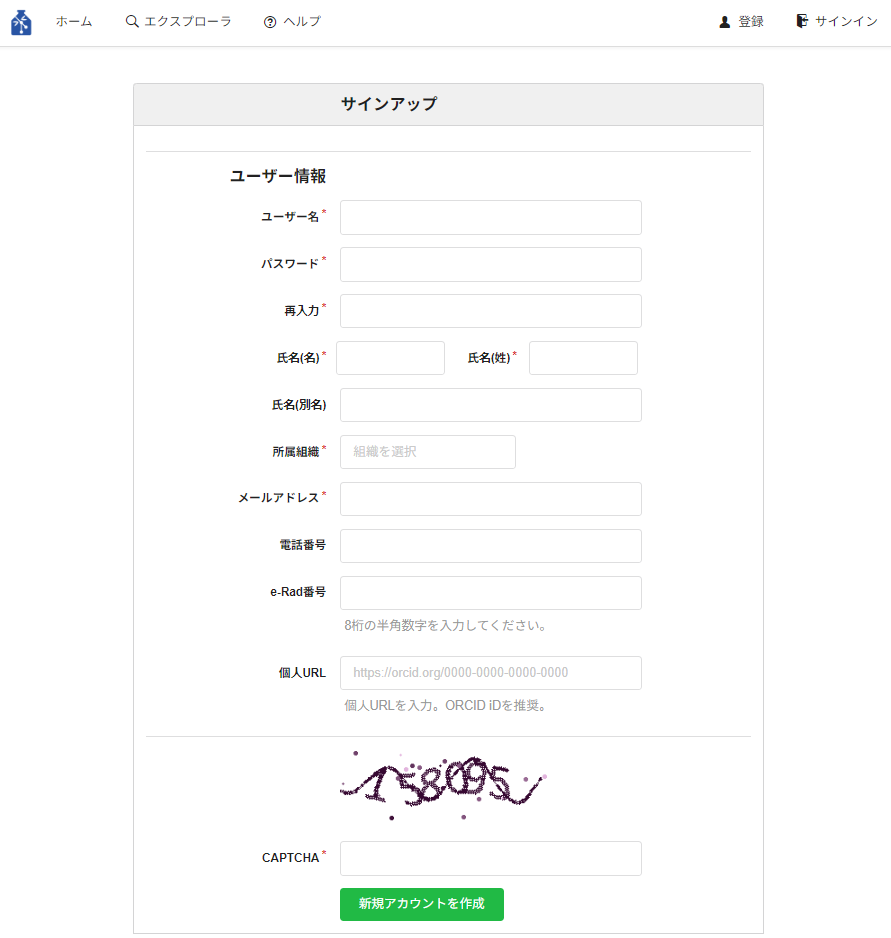
- 必要事項の入力
遷移したユーザー登録画面では、登録に必要な事項を入力していただきます。
それぞれの入力項目のうち、赤色のアスタリスク(*)がついている項目は必ず入力が必要です。それ以外は、入力の有無にかかわらず登録できます。
ユーザー情報は登録後にもユーザー設定画面にて変更ができます。
※ パスワードには、半角英数字記号が使用できます。スペースは使用できません。
※ 機能評価試験版では、個人URLにORCID iDを入力していない場合、「メタデータの検証」のタスクで「メタデータに不備が見つかりました」と表示されます。必要事項の入力が終わったら、下部の「新規アカウントを作成」ボタンを押下してください。これで登録は完了です。
- サインイン
ユーザー登録画面で「新規アカウントを作成」ボタンを押下すると、以下のようなサインイン画面へ遷移します。
登録いただいたユーザー名、パスワードを該当の欄に入力し、緑色の「サインイン」ボタンを押下してサインインしてください。
サインインに成功すると、あなたのダッシュボードが表示されます。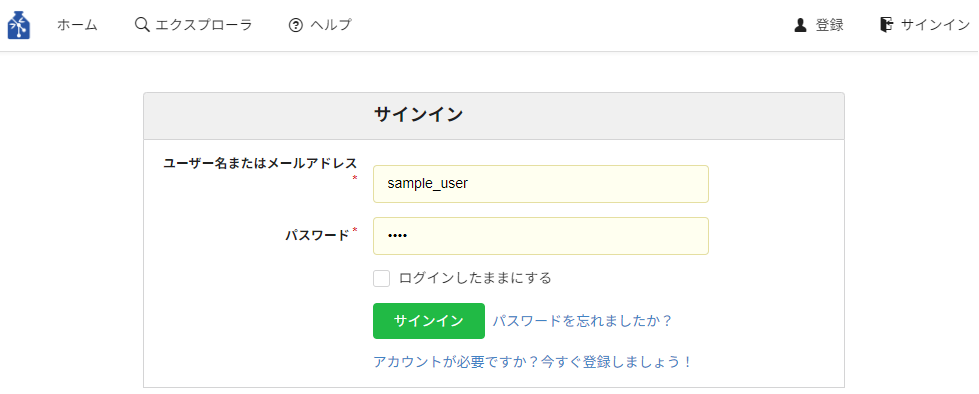
2. 研究を開始する
- 新しい研究を開始する
- 研究リポジトリの作成
新しく開始する研究プロジェクトのリポジトリを作成します。
GIN-forkのダッシュボード右側「自分のリポジトリ」の右端にある「+」をクリックすることで、リポジトリの作成を行えます。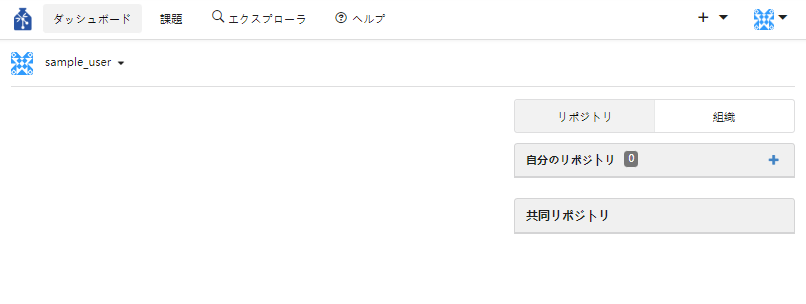
赤色のアスタリスク(*)がついている項目は必ず入力が必要です。それ以外は、入力の有無にかかわらず作成ができます。
リポジトリ名はアルファベット、数字、ハイフン(-)、アンダースコア(_) 、ドット(.) で入力してください。
リポジトリはデフォルトで非公開となっています。リポジトリを公開にする場合は「このリポジトリは非公開です」のチェックを外してください。
リポジトリ情報は作成後にもリポジトリ設定画面にて変更ができます。
入力完了後、「リポジトリを作成」ボタンを押下してください。
研究リポジトリを新規作成すると、その研究リポジトリのREADME.mdが表示されます。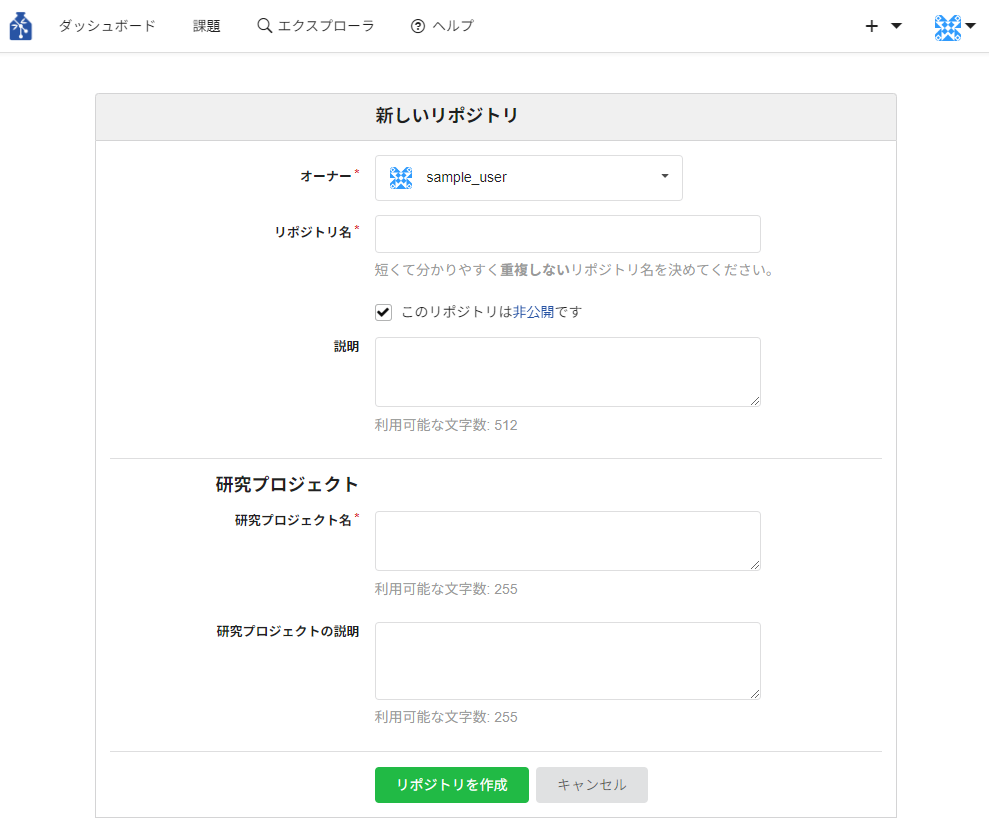
- DMPを登録
DMPの登録を行います。
画面右上の「Add DMP」からDMPの提出先を選択してください。
「Add DMP」は、DMPの種類を選ぶことができるプルダウンになっています。
システムで作成したDMPを実際に提出することはありませんが、任意の提出機関を選択してDMPを登録してください。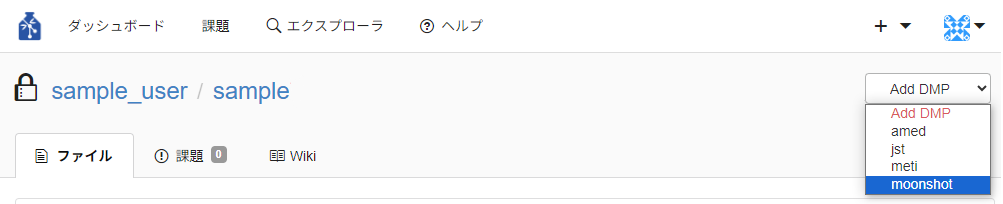
以下は選択できるDMPの種類です。
- amed : 国立研究開発法人日本医療研究開発機構(AMED) のDMP
- jst : 国立研究開発法人科学技術振興機構(JST) のDMP
- meti : 経済産業省(METI) のDMP
- moonshot : 内閣府が定めている公的資金による研究データの管理・利活用におけるメタデータの共通項目
提出先を選択すると、DMPの編集画面に遷移します。
DMPの編集画面で必要な情報を入力してください。選択したDMPの種類については、DMPの項目の「schema」の値をご覧ください。
DMPに記述する項目については、以下をご覧ください。■ GIN-forkで入力するDMPの項目について
本機能で作成するDMPには以下の種類が用意されています。各DMPのそれぞれの項目については、各機関の説明をご確認ください。
- amed : https://www.amed.go.jp/koubo/datamanagement.html
- jst : https://www.jst.go.jp/erato/manual/index.html
- meti : https://www.meti.go.jp/policy/innovation_policy/datamanagement.html
- moonshot : https://www8.cao.go.jp/cstp/kenkyudx.html
また、本機能では各研究機関で定められている記載項目だけでなく、本機能で独自に活用する情報も入力していただきます。
データガバナンス機能で活用する情報の説明は以下の通りです。- workflowIdentifier
使用するリサーチフローのテンプレートを選択してください。デフォルトは全分野で共通のテンプレートであるbasicです。
※ 現状では分野を選択してもbasicのテンプレートが使用されます。今後、各分野のテンプレートが増えていく予定です。 - contentSize
使用するデータ量を選択してください。検証機能で選択したデータ量を越えていないか確認できます。
- datasetStructure
実験で使用するパッケージの構造を選択してください。実験パッケージの種類については『実験パッケージについて』をご確認ください。
デフォルトではwith_codeが選択されています。
実験の初期セットアップで、選択したデータセット構造の実験パッケージが自動で作成されます。
設定した構造が保たれているかどうかは検証機能で確認できます。 - useDocker
データガバナンス機能では、実行環境の作成方法として「Dockerfile」による作成手段が提供されています。
必ずYESを選択してください。
※ 今後、他の方法による実行環境の作成を提供予定です。
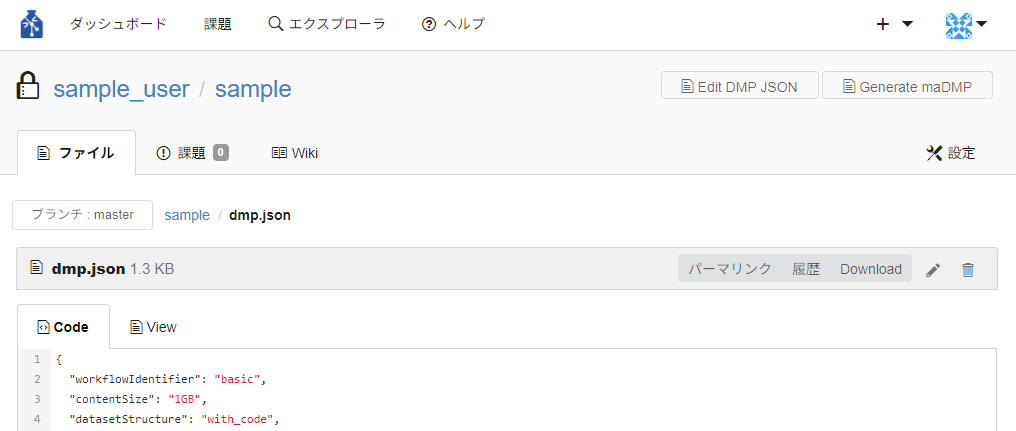
入力が完了したら、「変更をコミット」ボタンを押下してください。押下後、dmp.jsonが作成され、ファイルの内容が表示されます。
作成したDMPを編集する際は「Edit DMP JSON」ボタンを押下してください。
※ dmp.jsonのファイル名・ファイルパスは変更しないでください。DMPの内容はリポジトリトップのREADME.mdの下に表示されます。こちらでは表形式で確認できます。
- maDMPの作成
※ maDMPの作成以降、DMPを編集してもリサーチフローに取り込まれません。DMPの内容を変更したい場合はリポジトリの作成から再実施してください。
画面右上の「Generate maDMP」ボタンを押下してください。
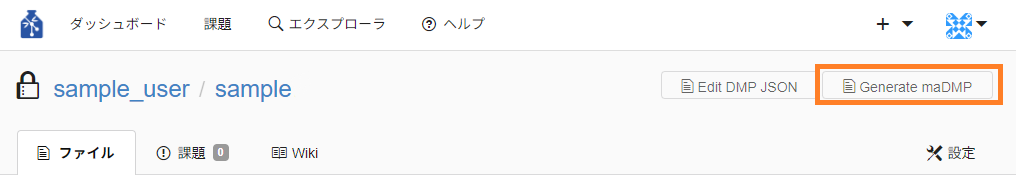
押下後、maDMPが作成され、実行環境タブへ遷移します。
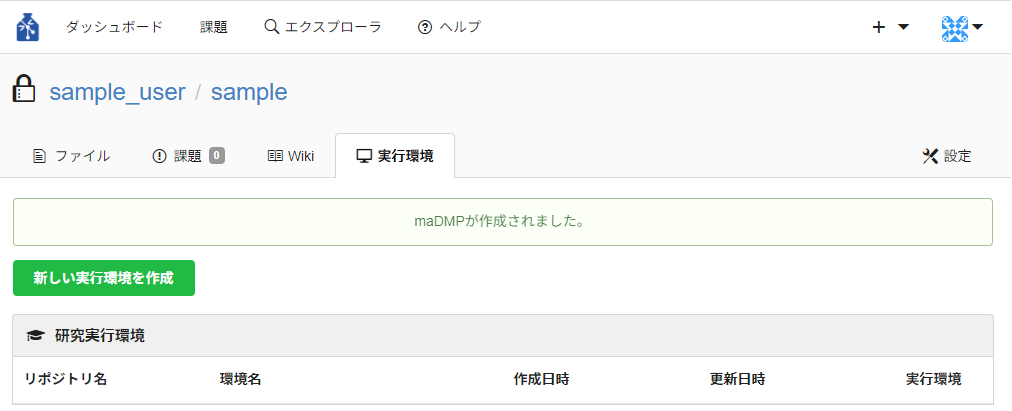
maDMPが作成されるとは以下のファイルがリポジトリに作成されます。
- maDMP.ipynb : コンピュータが実行可能なDMP
- Dockerfile : 環境構成ファイル
作成したmaDMPの詳細は画面右上にある「View maDMP」から確認できます。
- 環境構成ファイルのカスタマイズ(オプション)
インストールしたいパッケージがある場合はDockerfileに追記する必要があります。
以下の注意事項を守ってDockerfileを更新してください。
誤った更新を行うと正常なリサーチフローが提供できなくなる恐れがあります。■ 注意事項
① Dockerfileには、ユーザー独自の記述ができるエリア(ユーザーカスタムエリア)を用意しています(以下画像参照)。
ユーザーカスタムエリアのStratとEndの間に記述するようにしてください。② ユーザーカスタムエリア外の 既存の記述コードは編集しないでください。

- 研究実行環境へ遷移
実行環境タブの「新しい実行環境を作成」ボタンを押下してください。
非公開リポジトリの場合は実行環境を作成時にパスワードが必要となるため、パスワード入力画面へ遷移します。
GIN-forkサインイン時のパスワードを入力し「実行環境を作成する」のボタンを押下してください。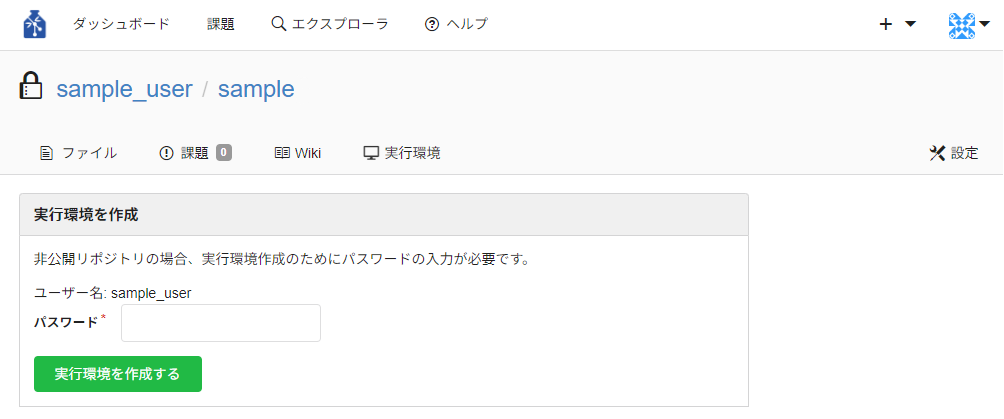
ボタンを押下すると、実行環境の作成が開始されます。
※ この処理には時間がかかります。
※ ログイン画面が表示された場合はデータ解析機能を利用できるアカウントでログインしてください。
作成に成功すると、自動的にmaDMP.ipynbが表示されます。
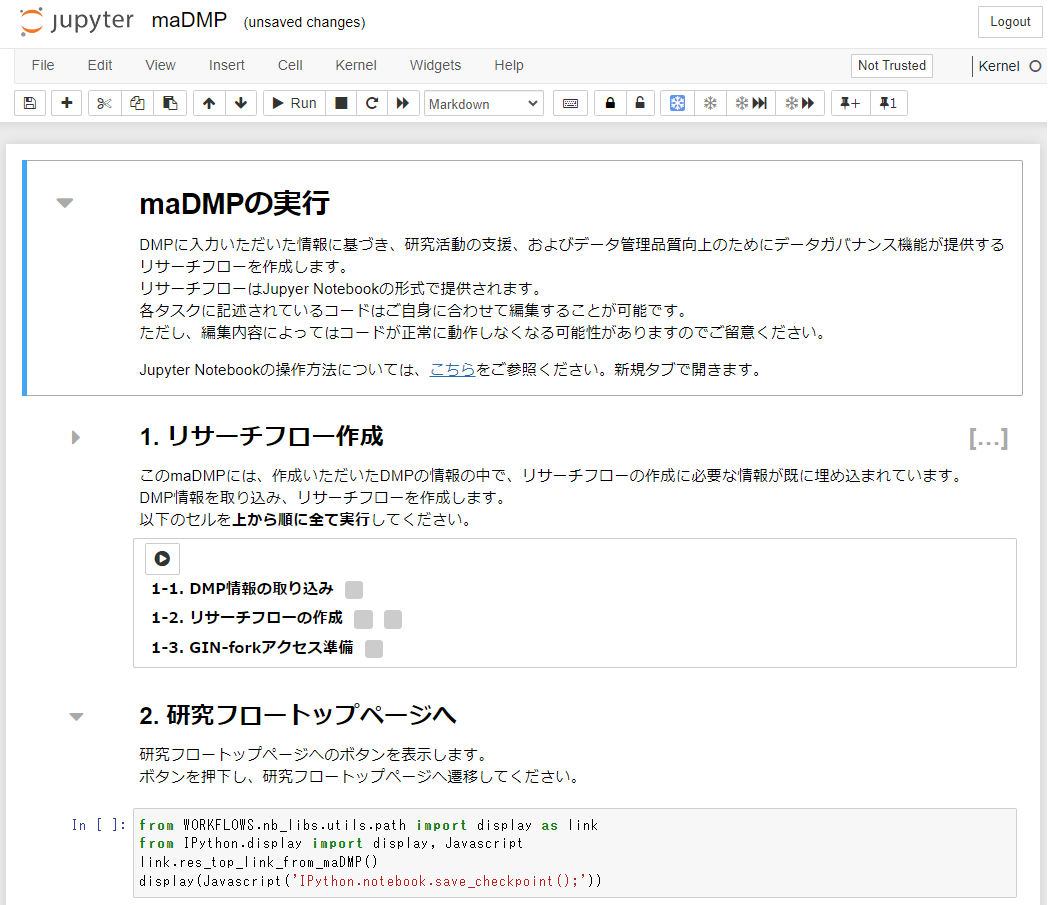
※ 作成時にエラーになってしまった場合は、『(先行利用)データガバナンス機能Q&A集』を確認してください。
※ 実行環境が維持されるのは最後に利用した日から30日間です。
※ 実行環境が消えてしまった場合は、実行環境タブの研究実行環境エリアから「実行環境作成」ボタンを押下し作成し直してください。
※ 作成できる実行環境は10個までです。 - maDMPの実行
maDMPは、コンピュータが実行可能な形式にDMPを変換したものです。
データガバナンス機能ではmaDMPをJupyter Notebook形式で半機械的に実行可能な手順書として提供しており、ここではDMPの情報からリサーチフローの作成を行います。作成されたリサーチフローもJupyter Notebook形式の手順書(タスクNotebook)で提供されます。
リサーチフローの各タスクに記述されているコードはご自身に合わせて編集することが可能です。
ただし、編集内容によってはコードが正常に動作しなくなる可能性がありますのでご留意ください。maDMP.ipynbの説明を読みながら、手順に従いセルを実行してください。
セルの実行については『付録2 Jupyter Notebookについて』をご確認ください。
全てのセルを実行後、「研究フロートップページへ」のボタンが表示されます。
ボタンを押下して遷移してください。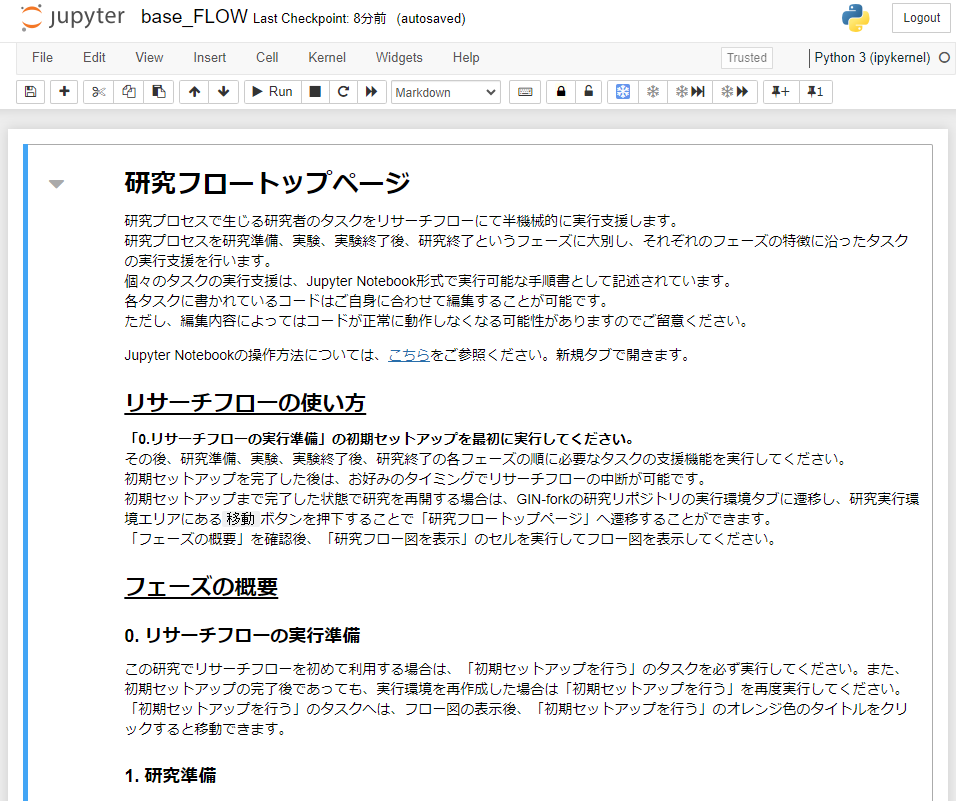
- 研究の初期セットアップ
研究実行環境の初期セットアップを行います。
「研究フロートップページ」に遷移後、「研究フロー図を表示」のセルを実行し、研究フロー図を表示してください。
研究フロー図の「0. リサーチフローの実行準備」フェーズの「初期セットアップを行う」をクリックしてタスクNotebookに遷移します。※ フロー図の各タスクのオレンジ色のタイトルをクリックすると、そのタスクのタスクNotebookに遷移することが出来ます。
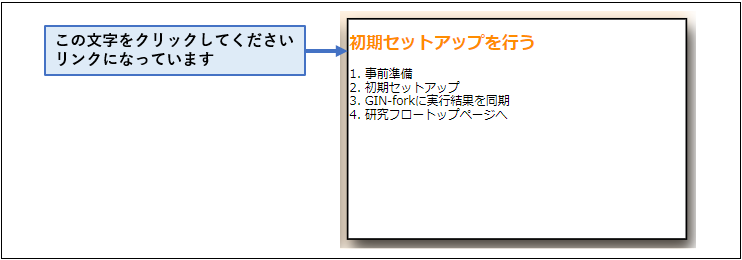
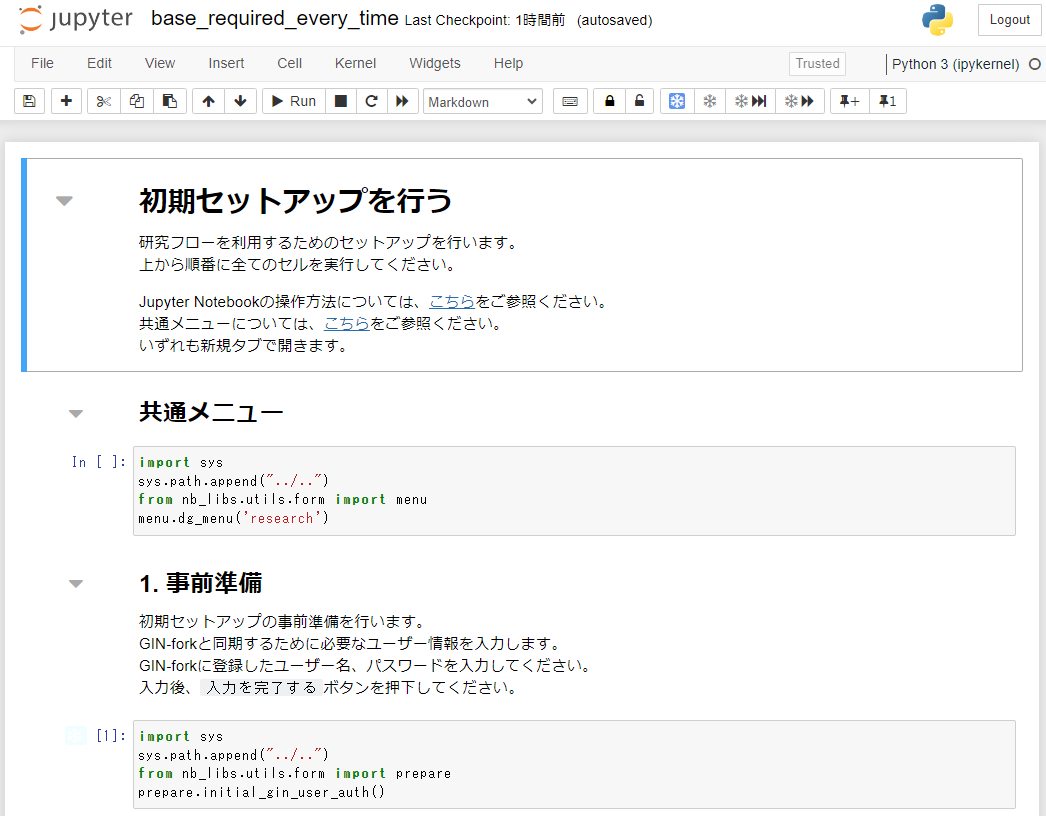
タスクNotebookの手順に従いセルを実行し、初期セットアップを完了させます。
初期セットアップ完了後、システムで使用するため以下のファイルやフォルダが作成・更新されます。
これらのファイルやフォルダの削除・更新等は行わないでください。- .gitignore : GIN-forkで管理しないファイルを定義したファイル
- WORKFLOWS : 各種フローを実行するためのファイルを格納したフォルダ
- README.md : データガバナンス機能の制限事項を記載
- maDMP.ipynb : コンピュータが実行可能なDMP
※ 「初期セットアップを行う」のタスクNotebookの全てのセルを実施せずに途中でNotebookを閉じてしまった場合は、『5.研究実行環境へ遷移』から再実施してください。
- 研究リポジトリの作成
- 研究を再開する(研究フロートップページを開く)
研究リポジトリを作成し、初期セットアップまで完了している場合は、実行環境タブの研究実行環境エリアより研究を再開することができます。
GIN-forkにサインインし、対象の研究リポジトリの実行環境タブに遷移してください。
研究実行環境エリアの実行環境より「移動」ボタンを押下して「研究フロートップページ」へ遷移します。「研究フロートップページ」に遷移できない場合は、研究実行環境エリアの「実行環境作成」のボタンを押下して研究実行環境の再作成を行ってください。
再作成後、『7. 研究の初期セットアップ』を再実施してください。
3. 実験を開始する
- 新しい実験を開始する
- 実験実行環境の作成
実験実行環境を作成します。
研究フロー図の「2. 実験」フェーズの「実験実行環境を作成する」をクリックしてタスクNotebookに遷移してください。遷移先の手順に従いセルを実行します。
セルの実行後、表示された「実験実行環境を作成」のボタンを押下して実験用の実行環境を作成します。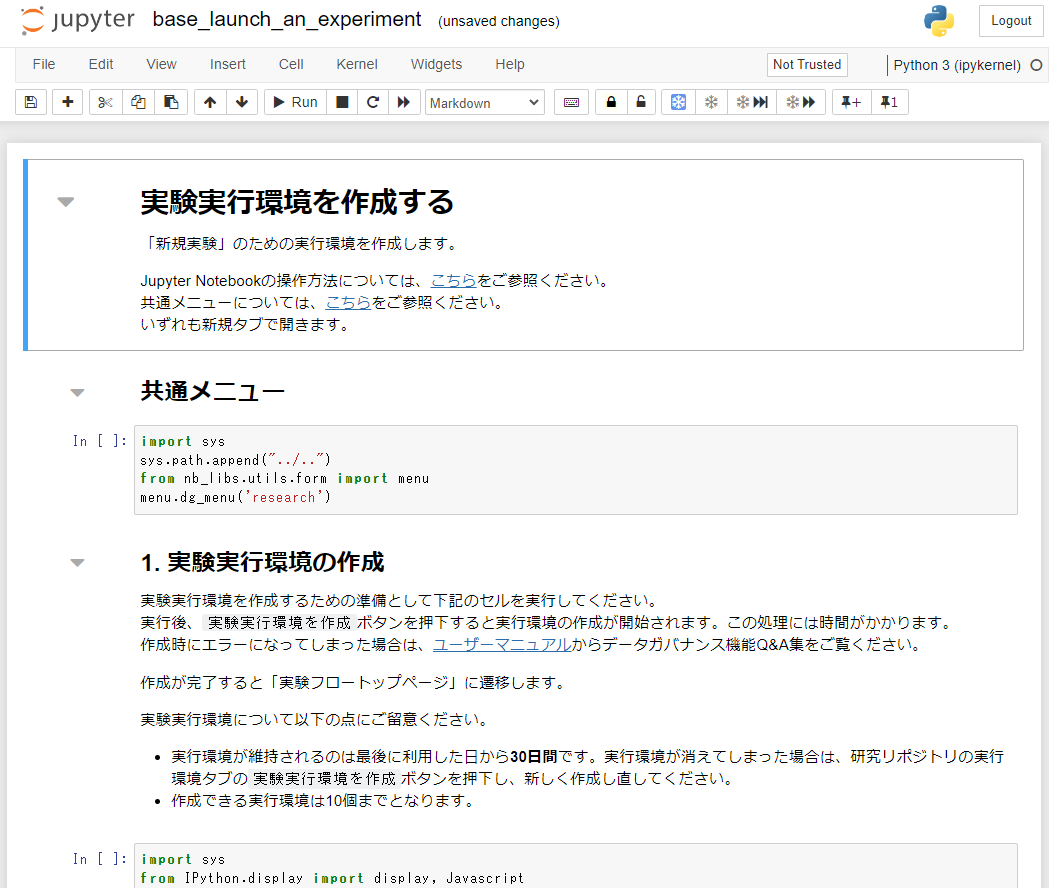
ボタンを押下すると、実行環境の作成が開始されます。
※ この処理には時間がかかります。
※ ログイン画面が表示された場合はデータ解析機能を利用できるアカウントでログインしてください。

成功すると、「実験フロートップページ」のタスクNotebookに遷移します。
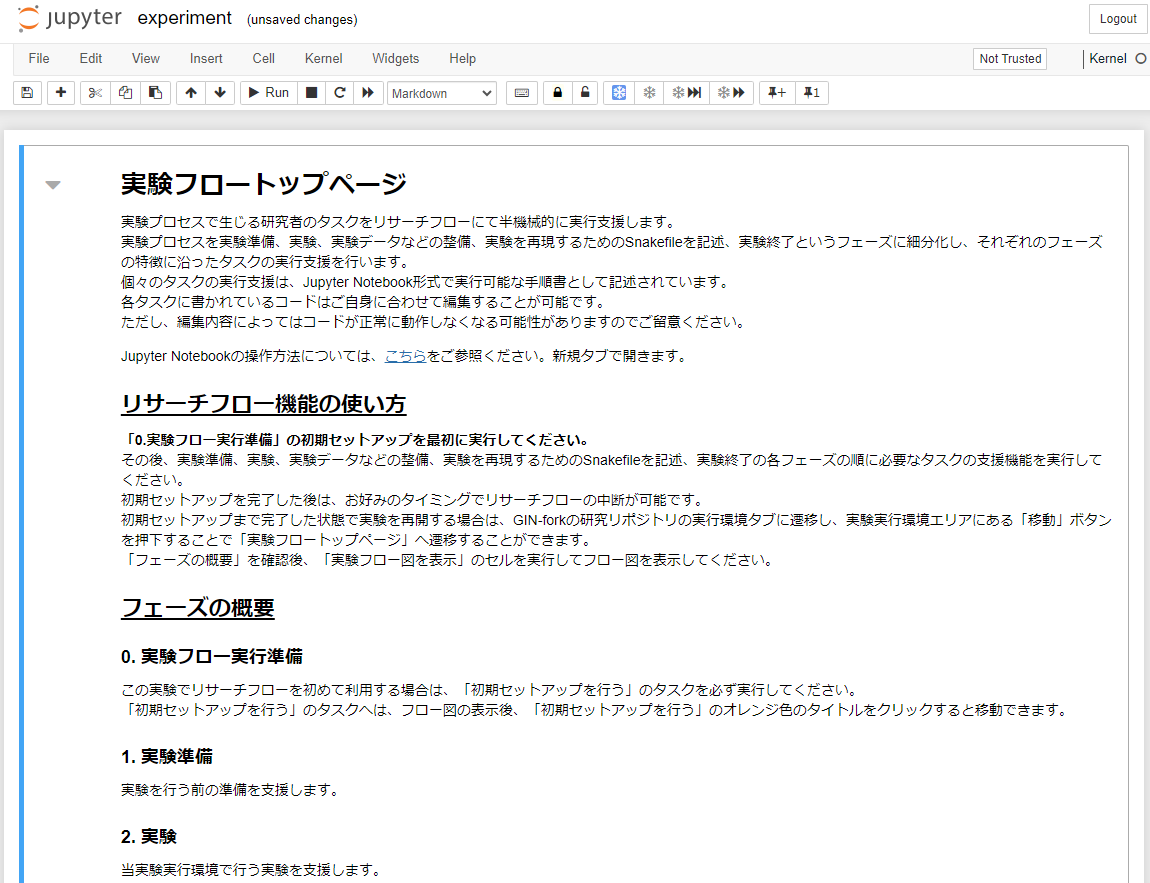
実験フロートップページへ遷移後は、『2. 実験の初期セットアップ』の最後の処理まで続けて実施してください。
途中でNotebookを閉じてしまった場合は「実験実行環境を作成」のボタンを押下するところから再実施が必要です。※ 作成時にエラーになってしまった場合は、『(先行利用)データガバナンス機能Q&A集』を確認してください。
※ 実行環境が維持されるのは最後に利用した日から30日間です。
※ 実行環境が消えてしまった場合は、実行環境タブの実験実行環境エリアから「実行環境作成」ボタンを押下し作成し直してください。
※ 作成できる実行環境は10個までです。 - 実験の初期セットアップ
実験実行環境の初期セットアップを行います。
「実験フロートップページ」に遷移後、「実験フロー図を表示」のセルを実行し、実験フロー図を表示してください。
実験フロー図の「0. 実験フロー実行準備」フェーズの「初期セットアップを行う」をクリックしタスクNotebookに遷移します。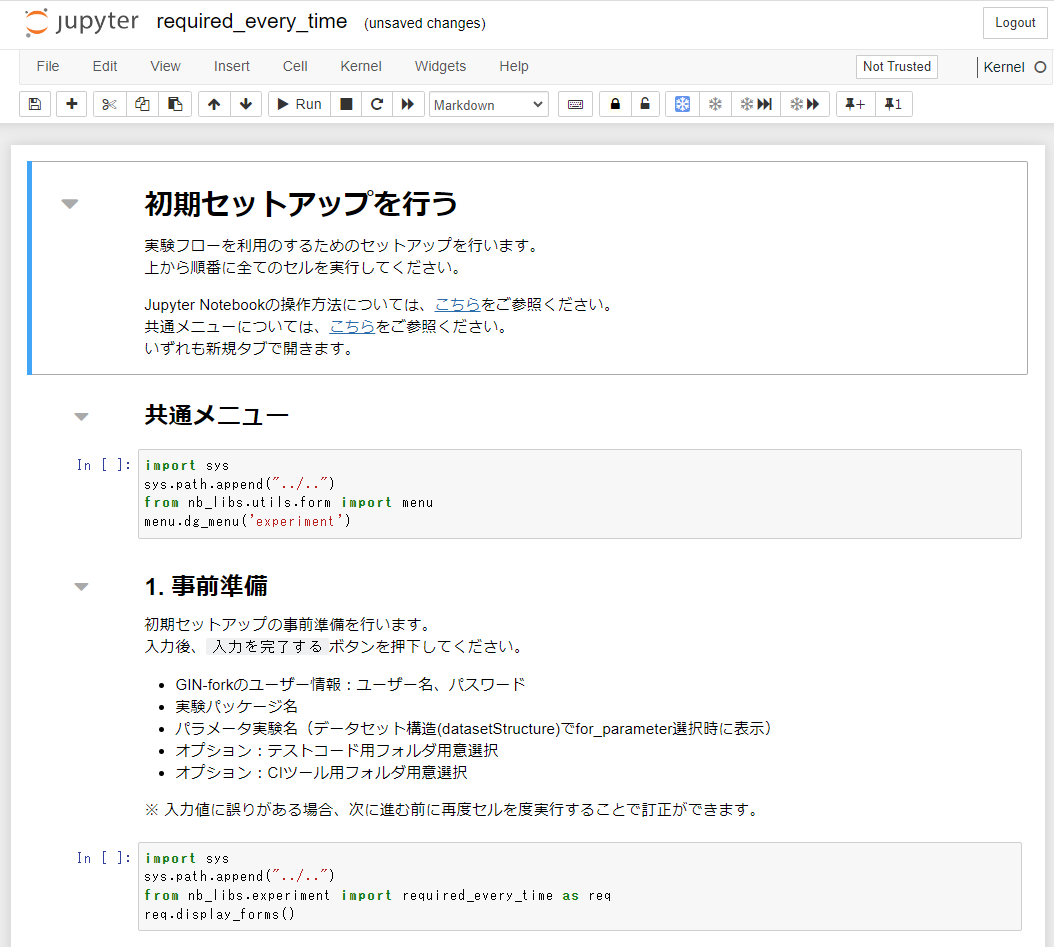
遷移後、初期セットアップの事前準備として以下の情報を入力してください。
- GIN-forkのユーザー情報
GIN-forkと同期するための準備として、あなたのユーザー情報をシステムに認証させる手続きを行います。
GIN-forkに登録したユーザー名、パスワードを入力してください。 - 実験パッケージ名
作成したい実験パッケージ名を50文字以内、半角英数字、-(ハイフン)、_(アンダーバー)、.(ドット)で入力してください。
研究リポジトリ内にすでに存在するパッケージ名は入力いただけません。実験名など分かりやすい名前をご記入ください。 - パラメータ実験名(データセット構造(datasetStructure)をfor_parameter選択時)
データセット構造(datasetStructure)として、with_codeを選択した場合は、表示されません。
データセット構造(datasetStructure)として、パラメータ用実験パッケ―ジ(for_parameters)を選択した場合、以下のような構造でデータが格納されパラメータ毎にoutput_dataとパラメータファイルを管理することができます。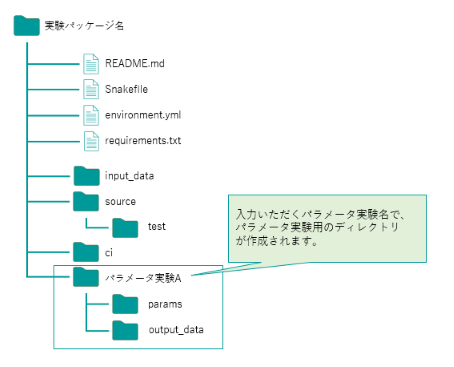
作成したいパラメータ実験名を50文字以内、半角英数字、-(ハイフン)、_(アンダーバー)、.(ドット)で入力してください。
パラメータ実験名は、実験パッケージ内で同名のパラメータ実験名を使用することはできません。
また、「parameter」というパラメータ実験名も使用することができません。パラメータ名など分かりやすい名前をご記入ください。 - オプション:テストコード用フォルダ用意選択
任意で選択するオプション項目です。
実験ソースコードのテストコードを作成する予定がある場合は選択してください。
ソースコードを格納する「source」ディレクトリ配下に、テストコードを格納するための「test」ディレクトリを用意します。
詳細は、『実験パッケージについて』を参照してください。 - オプション:CIツール用フォルダ用意選択
任意で選択するオプション項目です。
CI/CDツールを利用する予定がある場合は選択してください。
実験パッケージの直下にCI/CDツールの設定ファイル格納用の「ci」ディレクトリを用意します。
詳細は、『実験パッケージについて』を参照してください。
すべての項目の入力が完了後、タスクNotebookの手順に従いセルを実行し、初期セットアップを完了させます。
初期セットアップ完了後、システムで使用するため以下のフォルダが作成されます。 このフォルダの削除・更新等は行わないでください。
- experiments:実験パッケージを格納するフォルダです。初期セットアップで作成した実験パッケージが配下に格納されます。
■ 注意事項
実験パッケージを複数作成する場合は、研究フロートップページの「実験実行環境を作成する」のタスクを実行し、新しく実験実行環境を作成します。
その後、新しく作成した実験実行環境の実験フロートップページより「初期セットアップを行う」のタスクを実行し、実験パッケージを作成してください。
さらに実験パッケージを作成する場合は同様に実験実行環境を作成する所から繰り返してください。
同一実行環境で初期セットアップを実施して新しく実験パッケージを作成してしまうと、その実行環境は新しく作成した実験パッケージの実行環境となってしまいます。 - GIN-forkのユーザー情報
『1.新しい研究を開始する』の手順を全て完了していることが前提です。
- 実験実行環境の作成
- 実験の制約や進め方を確認する
実験を始める前に、実験の制約や進め方について確認します。
実験フロー図の「1. 実験準備」フェーズの「実験の制約や進め方を確認する」をクリックしてタスクNotebookに遷移してください。
遷移先にて実験パッケージの構造や実験の制約、進め方について確認することができます。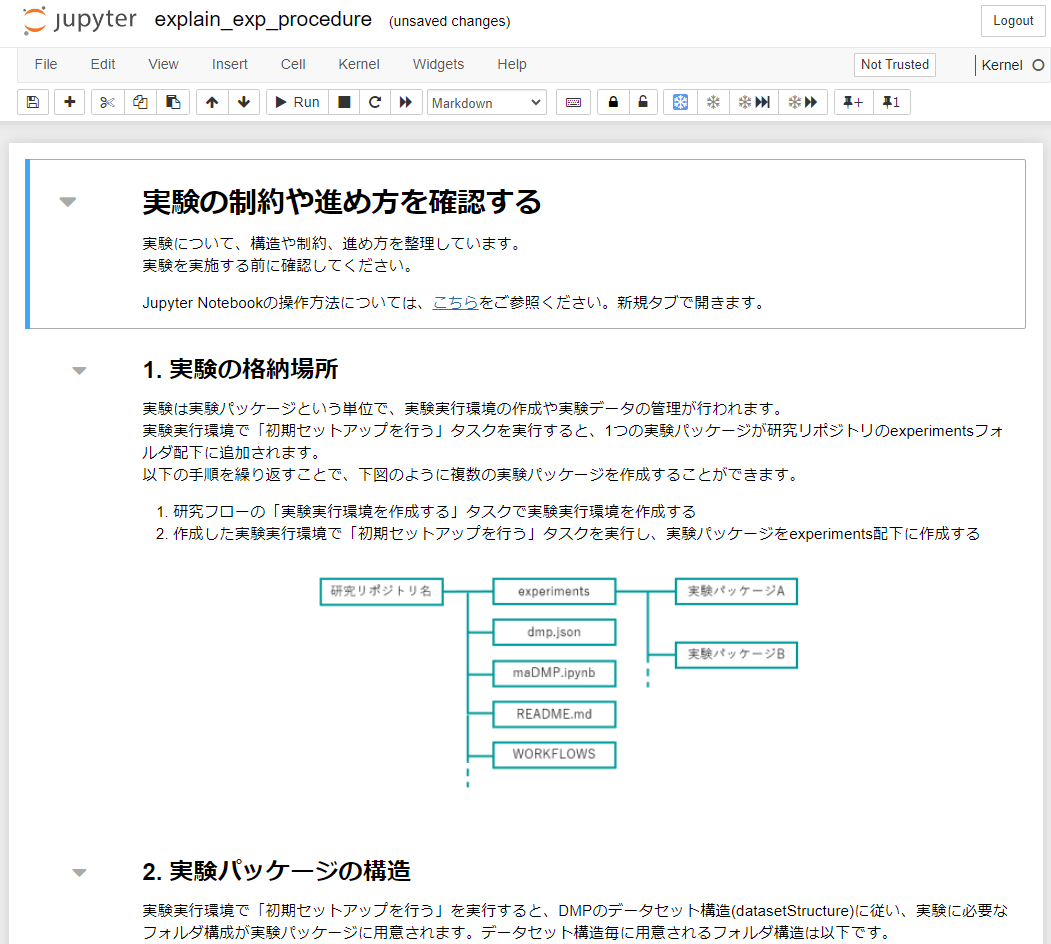
- 実験の入力データやソースコードを用意する
実験で必要な入力データやソースコードを用意するために外部やローカル環境からデータを取得することができます。
実験フロー図の「2. 実験」フェーズの「実験のデータやソースコードを用意する」をクリックしてタスクNotebookに遷移してください。
実験のデータやソースコードを用意する方法は以下となります。
- GIN-forkで公開しているリポジトリからデータを用意する
自分の他の研究リポジトリにある実験データや、他の研究者の研究リポジトリのデータを利用したい場合に実行してください。
※ 自分のリポジトリ、他の研究者のリポジトリ共に公開されているリポジトリのみ利用することができます。
非公開のリポジトリは利用できませんのでご留意ください。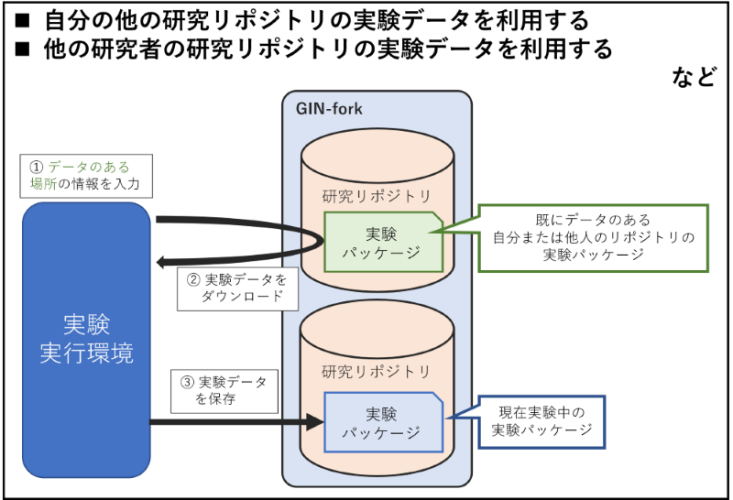
■ 研究リポジトリのURL取得方法
① 「エクスプローラ」タブをクリックします。

② 検索ボックスに対象のリポジトリ名を入力し、「検索」ボタンを押下します。表示された検索結果からリポジトリ名をクリックします。
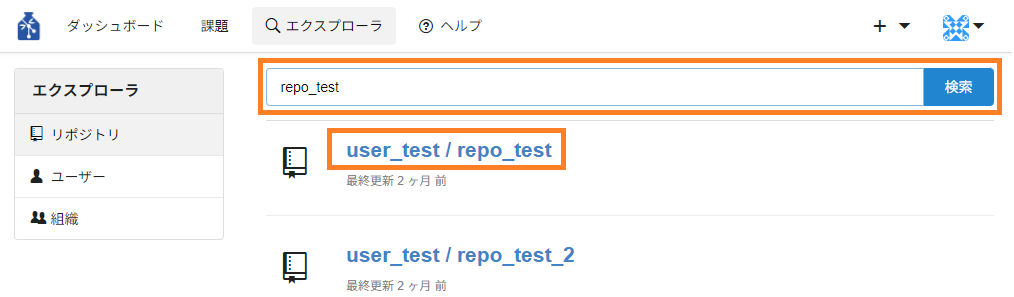
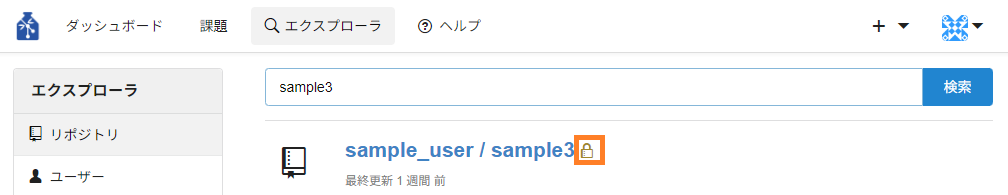
※ 鍵マークの付いているリポジトリは非公開のリポジトリとなります。利用ができませんのでご注意ください。
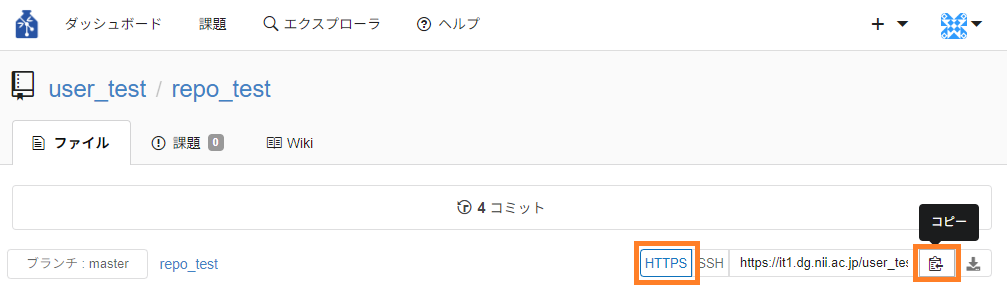
③ 対象のリポジトリのトップ画面で、HTTPSを選択し、URLをコピーします。
- S3からデータを用意する
自分または他の研究者などのS3のストレージにあるデータを、この実験の入力データや実験ソースコードとして利用したい場合に実行してください。
S3のデータを単一で取得するか、複数取得するかで取得方法が異なりますので、取得したいデータでいずれかを選択してください。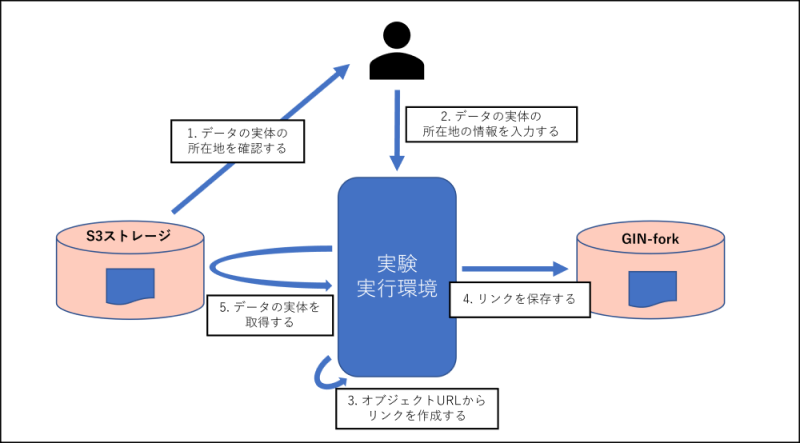
このタスクで用意できるS3ストレージのデータには、以下の制限があります。
実験のデータとして利用したいデータを格納したS3のバケット名にドット(.)を含めないでください。
また、このタスクは、パブリックにアクセスできるS3ストレージのデータを対象にしています。
そのため、非公開のデータは実験パッケージに用意できません。 - ローカル環境からデータを用意する
自分のローカル環境にあるデータを利用したい場合に実行してください。
■ 注意事項
① 取得したinput_data、output_data配下のファイルに対して編集を行いたい場合は、実験実行環境にて編集を行ってください。
git-annexにてファイル管理を行っているためGIN-forkから編集することができません。② ファイルの管理上、GIN-forkからinput_data、output_data配下にファイルを新規作成することはできません。
input_data、output_data配下にファイルを新規作成したい場合は、実験実行環境から行ってください。③ GIN-forkからファイルを追加・更新した場合、『4. 実験を途中保存する』の手順で実験実行環境と同期を行う必要があります。
ただし、並行して実験実行環境で操作をしていた場合、同期処理中に競合が発生してしまう場合があります。
競合が発生してしまった場合はタスクNotebook内で案内している「GIN-forkへの同期の失敗を解消する」を参照して、競合を解消してください。 - GIN-forkで公開しているリポジトリからデータを用意する
- パラメータ実験用のディレクトリを追加する
※ DMPのdatasetStructure項目でfor_parametersを選択した研究のみが対象のタスクです。
パラメータ実験用のディレクトリが複数必要な場合に、ディレクトリを追加することができます。
実験フロー図の「2. 実験」フェーズの「パラメータ実験用のディレクトリを追加する」をクリックしてタスクNotebookに遷移してください。
遷移先の手順に従い、パラメータ実験用のディレクトリを追加します。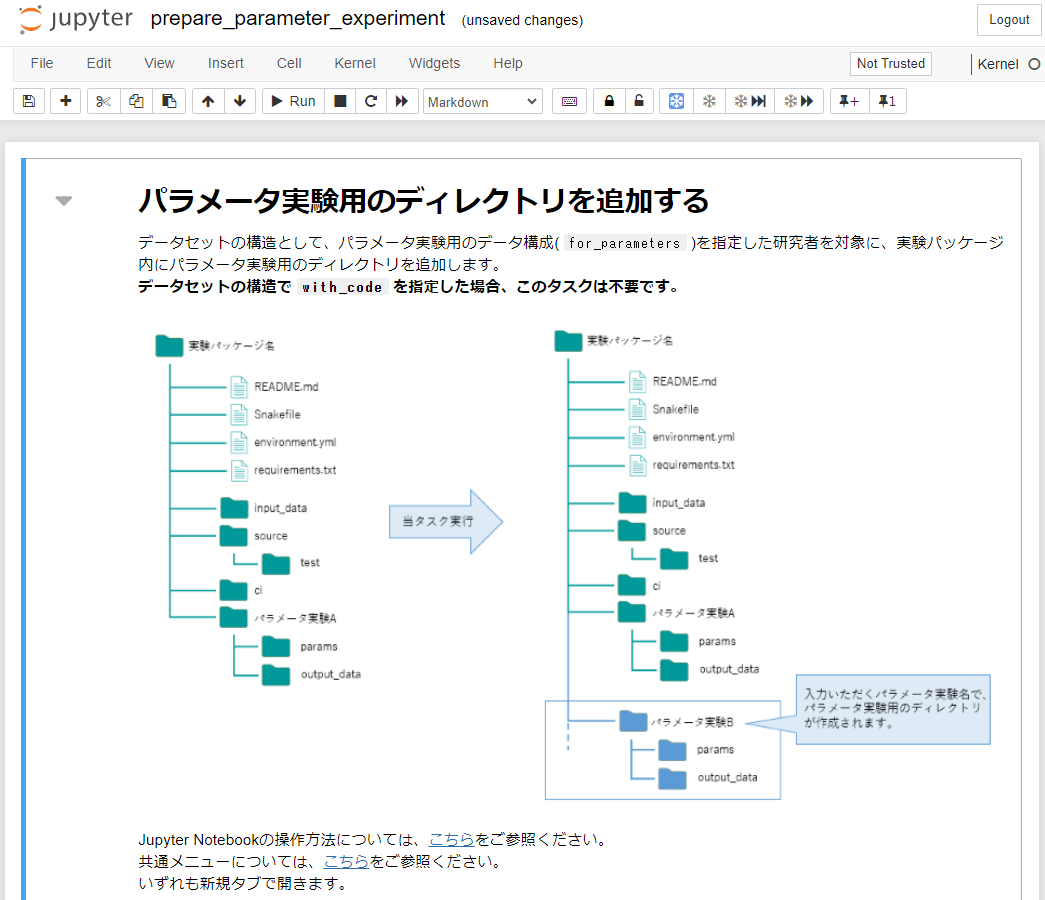
4. 実験を途中保存する
- 実験を途中保存する
実験実施において、実験実行環境での作業を中断する場合や編集中のデータ、実験途中の結果のデータなどを保存したい場合に実施してください。
実験フロー図の「2. 実験」フェーズの「実験の途中保存」をクリックしてタスクNotebookに遷移します。
遷移先の手順に従い、実験実行環境の実験パッケージ内の変更をGIN-forkに同期します。この操作を行わないと、GIN-forkに実験パッケージ内の変更が同期されません。
GIN-forkに同期していない場合、実験実行環境に問題が生じた際に実験を復元できませんのでご注意ください。※ GIN-forkのファイルに変更を行ったなどGIN-fork側を更新した場合もこのタスクを実施してください。実行実験環境に同期されます。
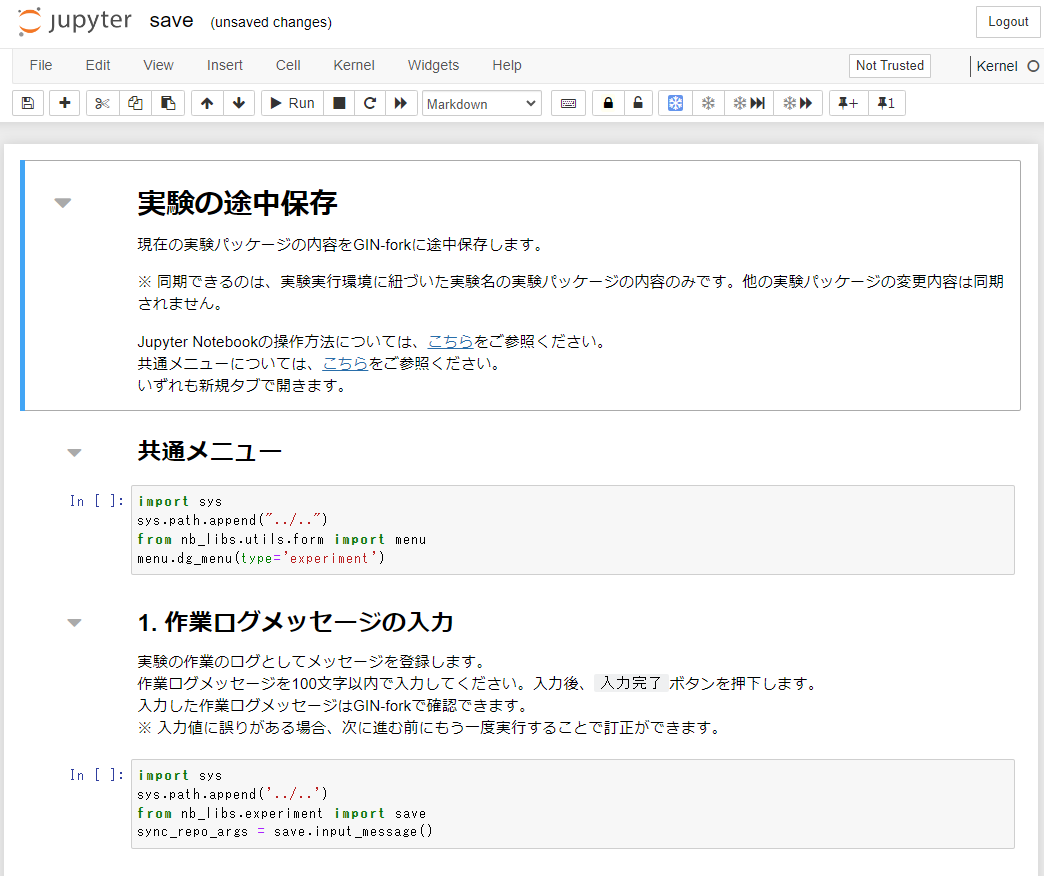
- 作業ログメッセージの確認方法
GIN-forkのリポジトリ画面にて、入力した作業ログメッセージが確認できます。また、コミット履歴に保存の履歴が残ります。
コミット履歴については『3.ファイルタブ』を参照してください。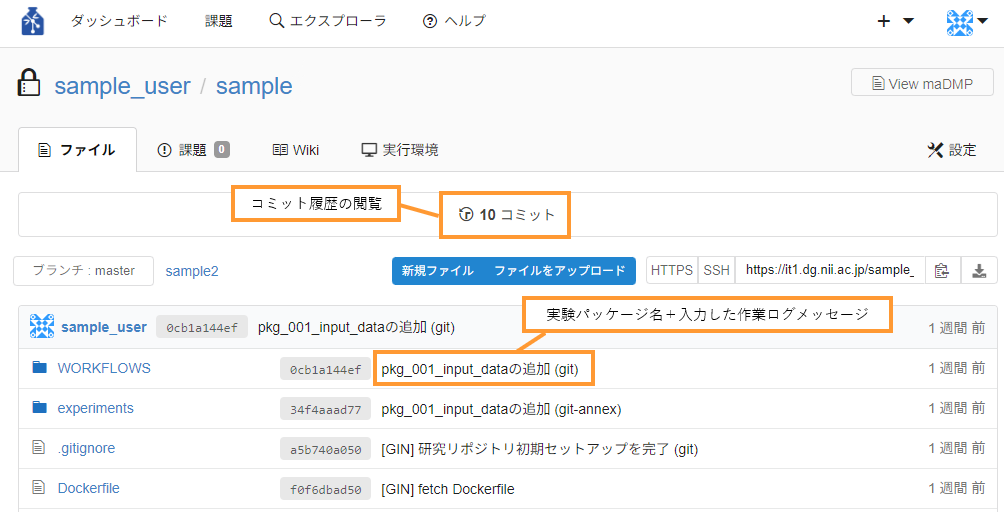
- 実験を再開する(実験フロートップページを開く)
実験フローの初期セットアップが完了している場合は、実行環境タブの実験実行環境エリアより実験を再開することができます。
GIN-forkにサインインし、対象の研究リポジトリの実行環境タブに遷移します。
実験実行環境エリアの実行環境より「移動」ボタンを押下して「実験フロートップページ」へ遷移します。初期セットアップが完了していない場合は、『1.新しい実験を開始する』の手順を最初から再実施してください。
初期セットアップは完了しているが「実験フロートップページ」に遷移できない場合は、実験実行環境エリアの「実行環境作成」のボタンから実験実行環境の再作成を行うことができます。
ボタンを押下すると「実験実行環境を再作成した場合のセットアップを行う」タスクNotebookに遷移します。
遷移先の手順に従い、実験実行環境の再作成を行ってください。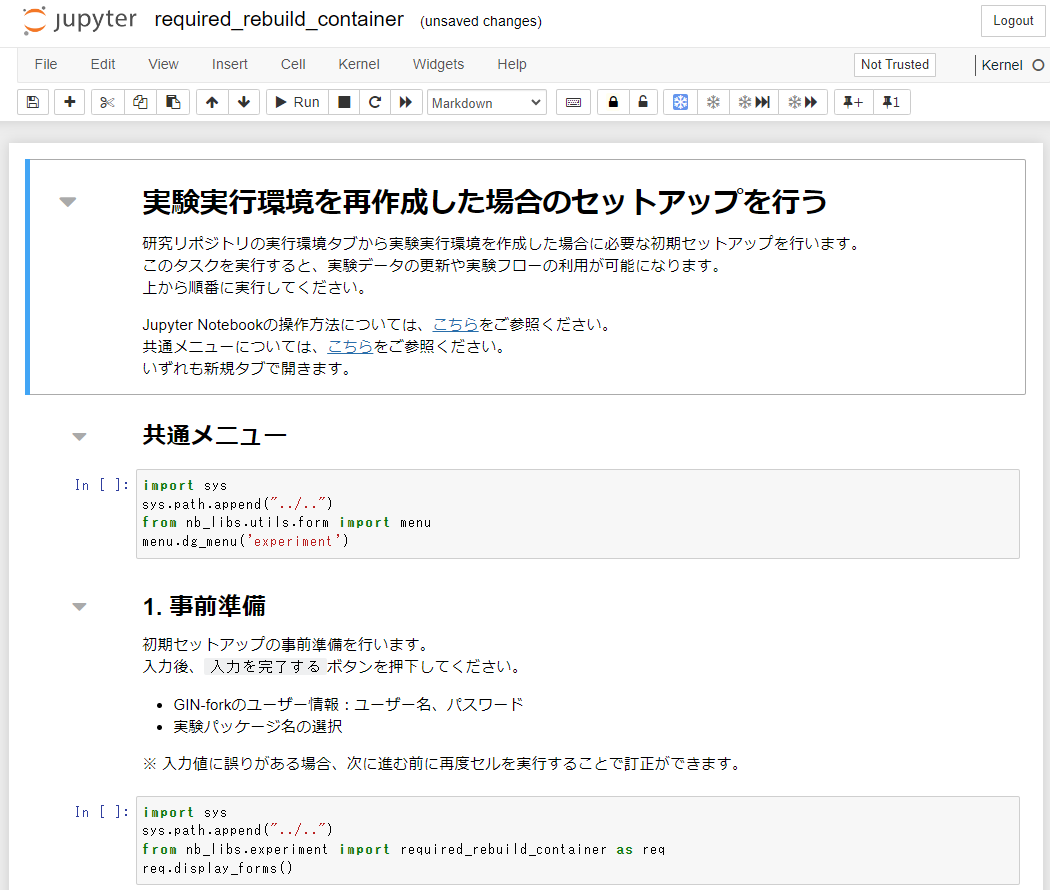
5. 実験の説明を記述する
実験についての説明を実験パッケージのREADME.mdに記述しGIN-forkに保存します。
実験フロー図の「3. 実験データなどの整理」フェーズの「実験の説明を記述する」をクリックしてタスクNotebookに遷移してください。
遷移先の手順に従い、実験の説明を実験パッケージのREADME.mdに記述します。記述後、GIN-forkに同期してください。
6. 実験を再現するためにSnakefile(タスクランナー)を記述する
実験パッケージの再現性を確保するために、実験内の処理の依存関係などの実験の実行方法をSnakefile(タスクランナー)に記述します。
実験フロー図の「4. 実験を再現するためのSnakefileを記述」フェーズの「Snakefileに実験手順を記述する」をクリックしてタスクNotebookに遷移してください。
遷移先の手順に従い、実験の手順を実験パッケージのSnakefileに記述します。記述後、GIN-forkに同期してください。
7. 実験を終了する
- 実験実行環境を削除する
実験を終了する場合、実験パッケージの内容をGIN-forkに同期して、実験実行環境を削除します。
実験フロー図の「5. 実験終了」フェーズの「実験を終了する」をクリックしてタスクNotebookに遷移します。
遷移先の手順に従い、実験実行環境を削除してください。※ 実行環境の停止・削除後は、実行環境にあるNotebookを操作することが出来なくなりますのでご注意ください。
- 終了した実験を再実施する
実験実行環境を削除した後に実験を再開したい場合は、実行環境を再作成する必要があります。
GIN-forkにサインインし、対象の研究リポジトリの実行環境タブに遷移してください。
実験実行環境エリアの「実行環境作成」のボタンを押下すると、「実験実行環境を再作成した場合のセットアップを行う」のタスクNotebookに遷移します。
手順に従い実験実行環境の再作成を行ってください。
8. メタデータを検証する
以下の点にご留意ください。
※ 実験実行環境の初期セットアップを終了していない場合、「メタデータを検証する」タスクでエラーが表示されます。
※ ユーザー情報の個人URLにORCID iDを入力していない場合、「メタデータを検証する」タスクで「メタデータに不備が見つかりました」と表示されます。
研究フロー図の「2. 実験」フェーズの「メタデータを検証する」をクリックしてタスクNotebookに遷移します。遷移先の手順に従いセルを実行してください。
検証結果は、記録をしてGIN-forkの研究リポジトリに同期するか、その場での確認のみで記録をせず破棄をするか選択することができます。
検証結果を破棄せずに記録をした場合は、以下の手順で検証結果を確認することができます。
- フォルダ構成
検証結果は研究リポジトリの直下のvalidation_resultsフォルダに格納されます。
validation_resultsフォルダの構成は以下です。
- 確認手順
① GIN-forkの検証結果を確認したいリポジトリトップ画面に遷移し、validation_resultsフォルダをクリックします。
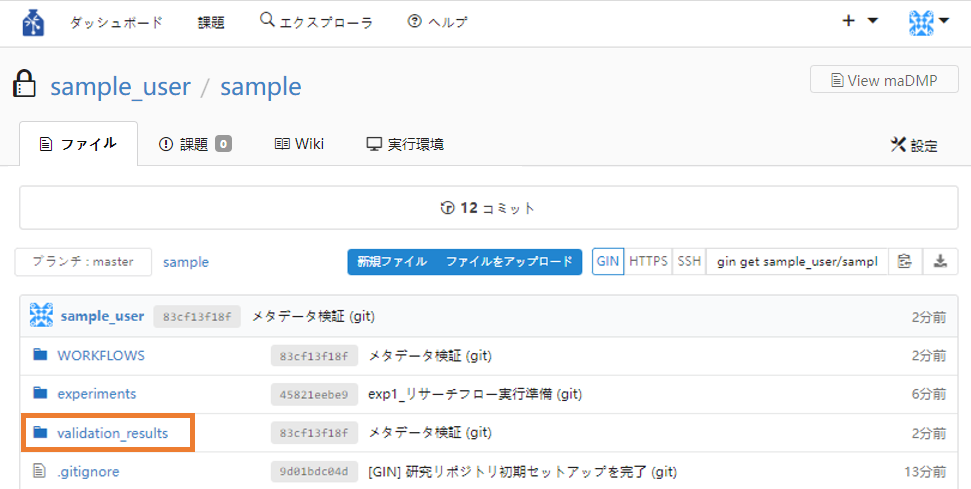
② results.jsonをクリックします。
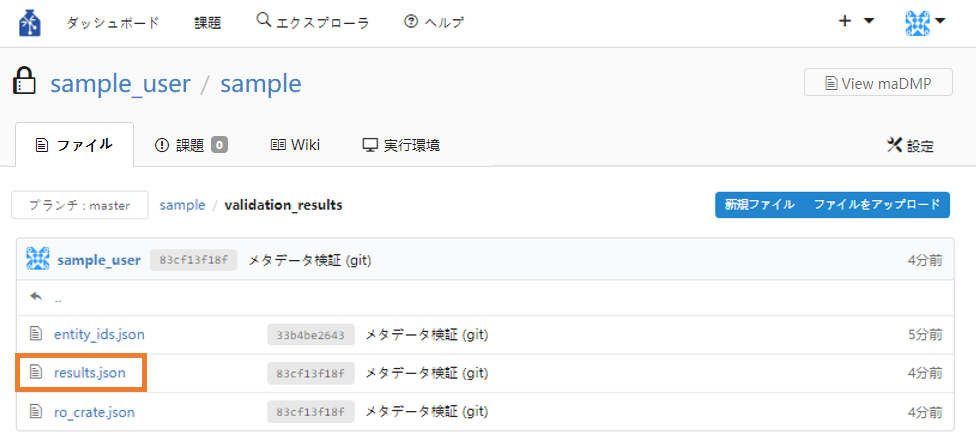
③ 最新の検証結果が表示されます。
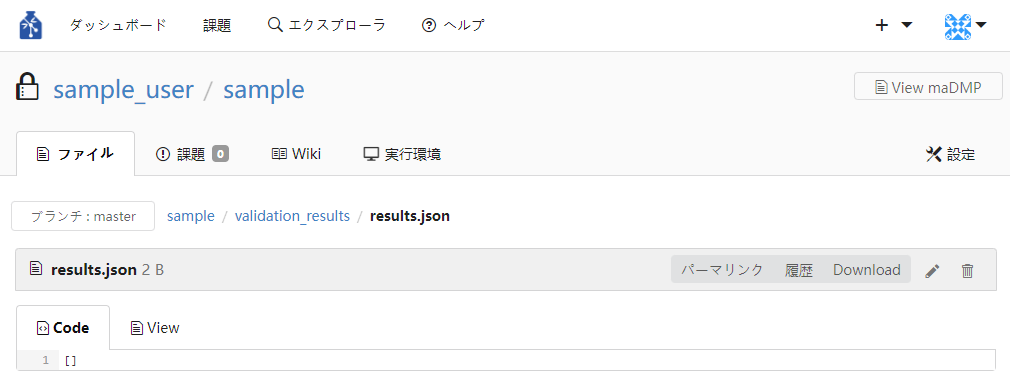
過去の検証結果を確認したい場合は、以下の手順を続けて行ってください。
④ 履歴をクリックします。
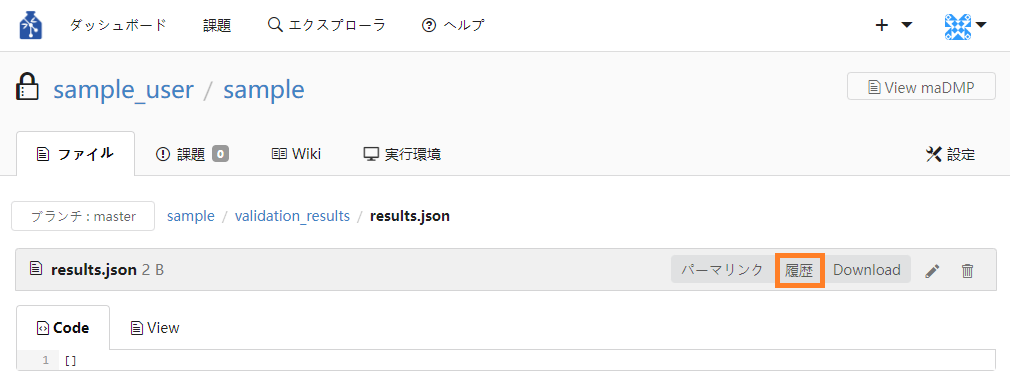
⑤ 検証結果の履歴が降順で一覧表示されるので、確認したいバージョンの行のSHA1のリンクをクリックします。
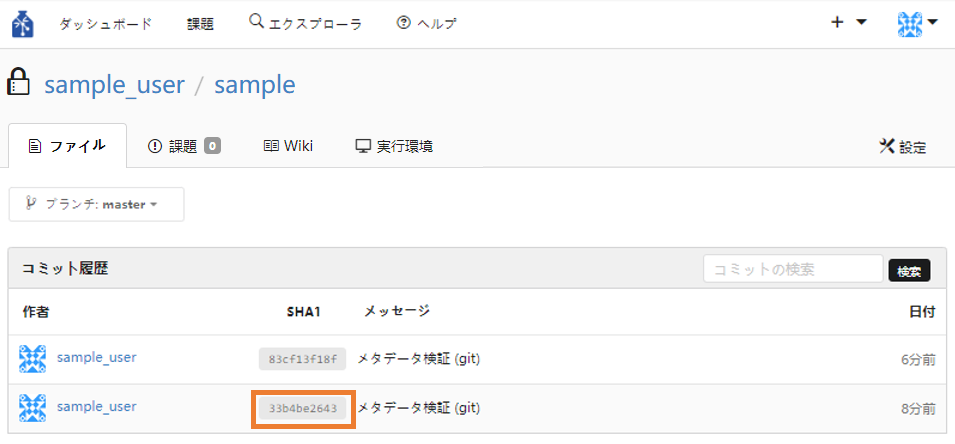
⑥ 右上のソースを参照をクリックします。
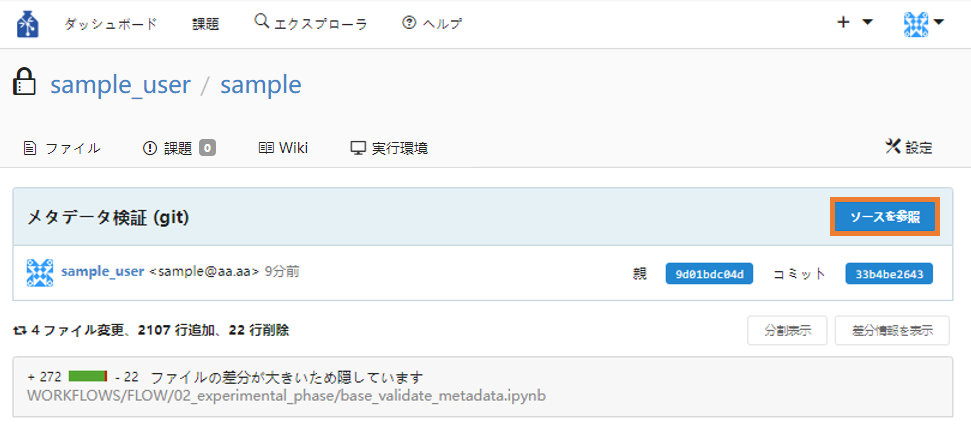
⑦ validation_resultsフォルダをクリックします。
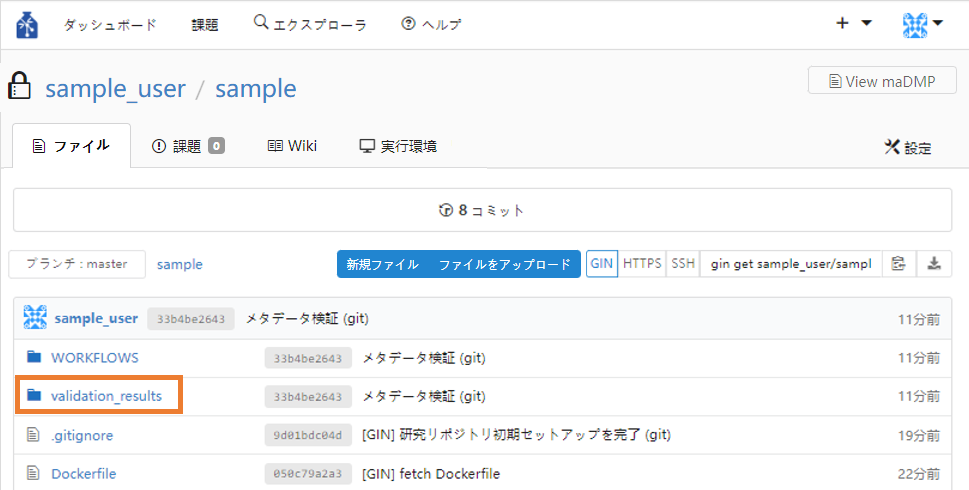
⑧ results.jsonをクリックします。
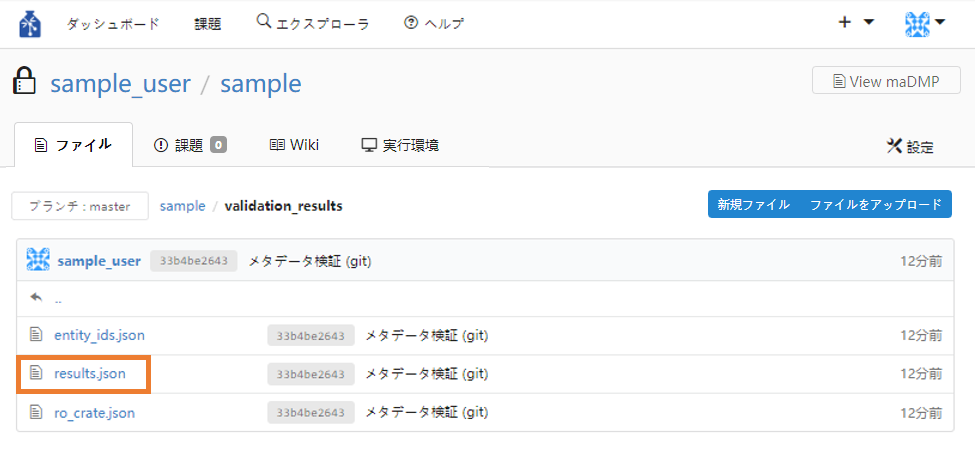
⑨ 選択したバージョンの検証結果が表示されます。
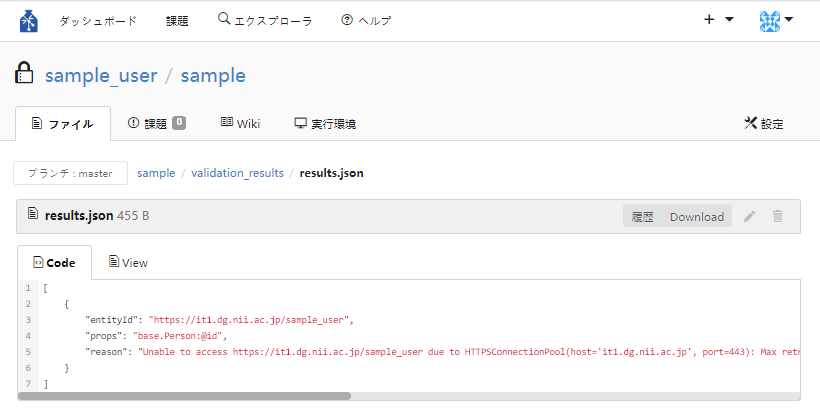
- 出力サンプル
validation_resultsフォルダ配下のファイルのサンプルは以下です。
- ro_crate.json
- entity_ids.json
- results.json
■ 全検証項目が検証OKの場合
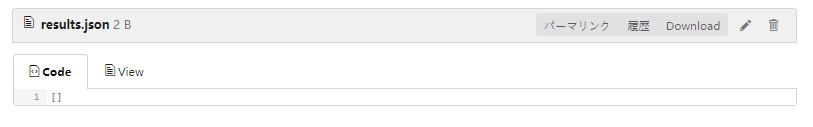
■ 検証NG項目がある場合
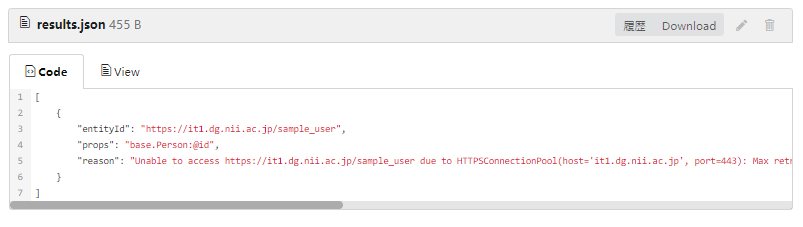
- ro_crate.json
9. 3~7を繰り返して研究を進める
10. 研究を終了する
研究結果の公開などを終えて研究を終了させる場合、使用していた研究実行環境を削除します。
研究フロー図の「4. 研究終了後」フェーズの「研究を終了する」をクリックしてタスクNotebookに遷移します。
遷移先の手順に従い、研究実行環境を削除してください。
※ 実行環境の停止・削除後は、実行環境にあるNotebookを操作することが出来なくなりますのでご注意ください。
11. 複数の研究を実施したい場合は2~9を繰り返す(並行実施可)
付録1 GIN-forkの操作について
- リポジトリトップ画面
リポジトリやファイルに対する操作を行うことができます。
ファイル名やフォルダ名をクリックすると、ファイルやフォルダを閲覧することができます。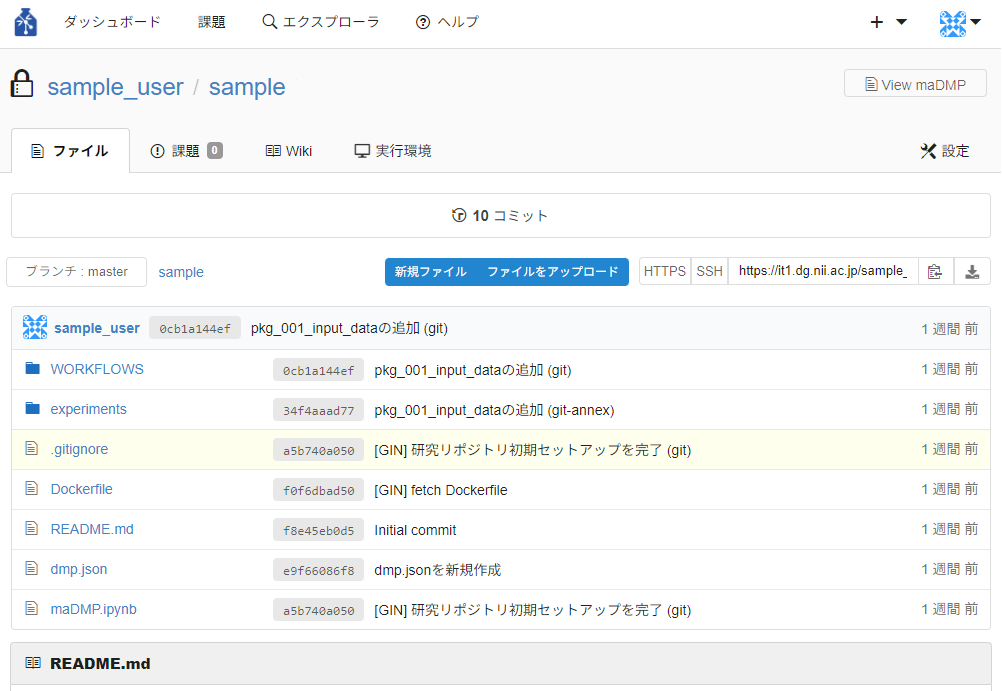
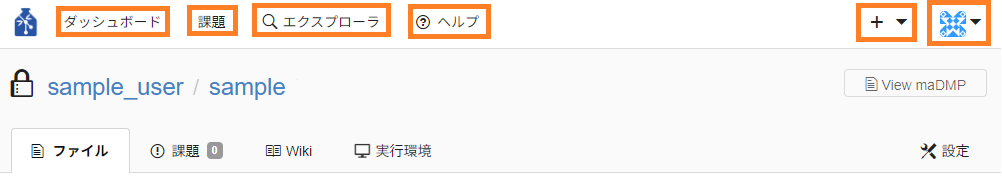
- ヘッダー
- ダッシュボード : 自分がリポジトリに対して行った操作を確認することができます。
- 課題 : ポジトリに対して作成された課題を確認することができます。
- エクスプローラ : 他ユーザーが作成したリポジトリなどを確認することができます。
- ヘルプ : データガバナンス機能のヘルプを確認することができます。
- +:新規にリポジトリか組織を作成することができます。
- ユーザーアイコン : ユーザーのアイコンをクリックし「プロフィール」をクリックすると、ユーザーページに遷移します。
「設定」をクリックすると、ユーザー設定画面に遷移します。
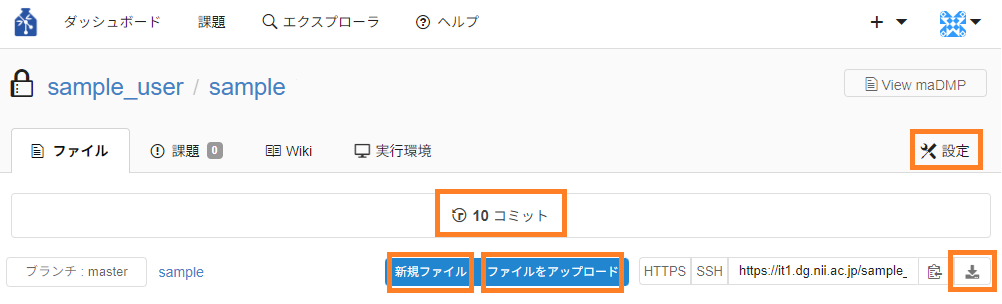
- ファイルタブ
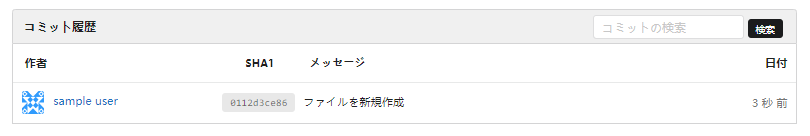
- コミット : コミットの回数が表示されます。クリックすると、リポジトリに対するコミット履歴画面に遷移します。
- 新規ファイル : ファイルを新規作成することができます。ファイル名は63文字までとなります。
- ファイルをアップロード : 自分のローカルからファイルをアップロードすることができます。
- 設定 : リポジトリ設定画面に遷移します。
- ダウンロード:リポジトリの全てのデータをzip形式またはtar.gz形式でダウンロードすることができます。
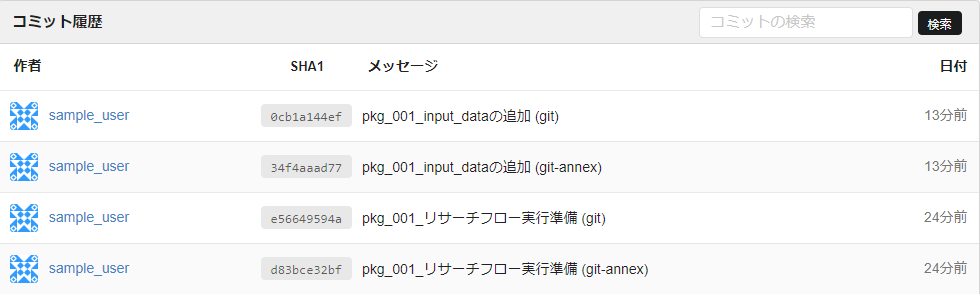
- コミット履歴 : コミット履歴画面ではコミットの履歴が表示されます。ハッシュ値(SHA1)やメッセージを確認することができます。
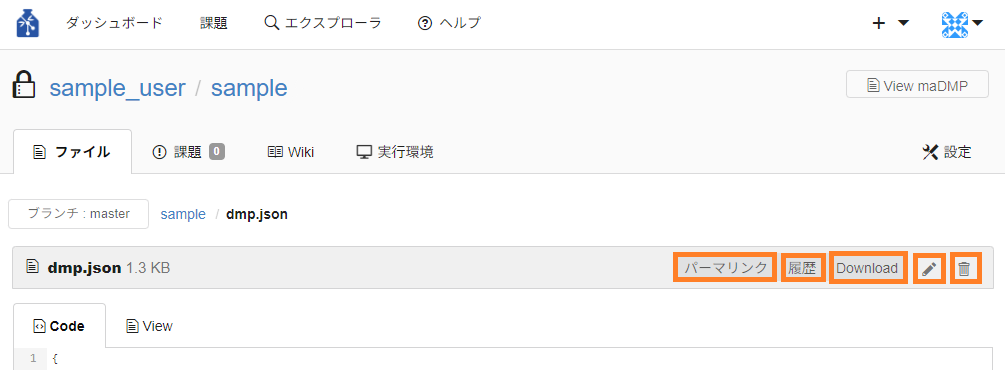
- ファイル閲覧画面
- パーマリンク : 現時点でのこのファイルに対する恒久的なリンクを取得することができます。
- 履歴 : ファイルに対する変更の履歴を確認することができます。
- Download : ファイルをダウンロードすることができます。
- 鉛筆マーク : テキストファイルの編集を行うことができます。
- ゴミ箱マーク : ファイルの削除を行うことができます。
- 外部から取得したデータ
リサーチフローの「実験のデータやソースコードを用意する」タスクで、GIN-forkの公開リポジトリやS3から用意したファイルの場合は、以下のように表示されます。
これらのファイルについては、データサイズが大きい場合が考えられるため、閲覧画面にファイルの内容が表示されませんが、「ファイルのコンテンツへアクセスする」をクリックすると、ファイルのコンテンツ(中身)をご確認いただけます。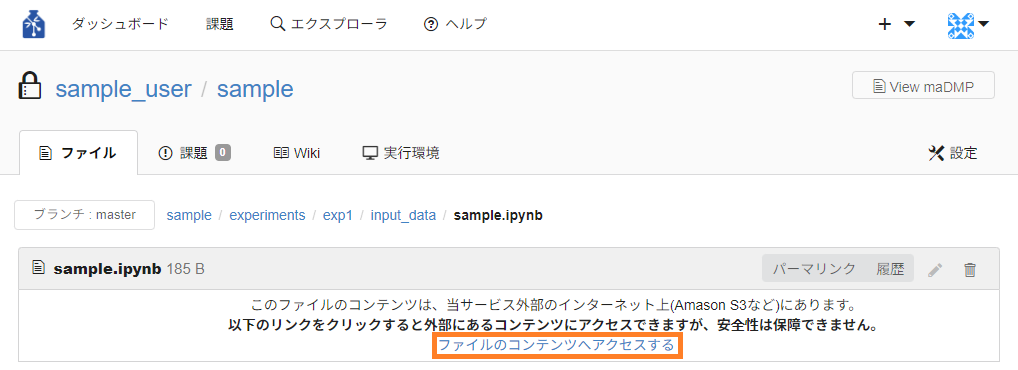
- 実行環境タブ
作成した研究実行環境と実験実行環境を一覧で確認をすることができます。
初期セットアップが完了している場合、各環境の「移動」ボタンを押下することで実行環境(研究フロートップページ、実験フロートップページ)へ遷移することができます。
環境を削除してしまった場合などに各環境の「実行環境作成」ボタンを押下することで各環境を再作成することができます。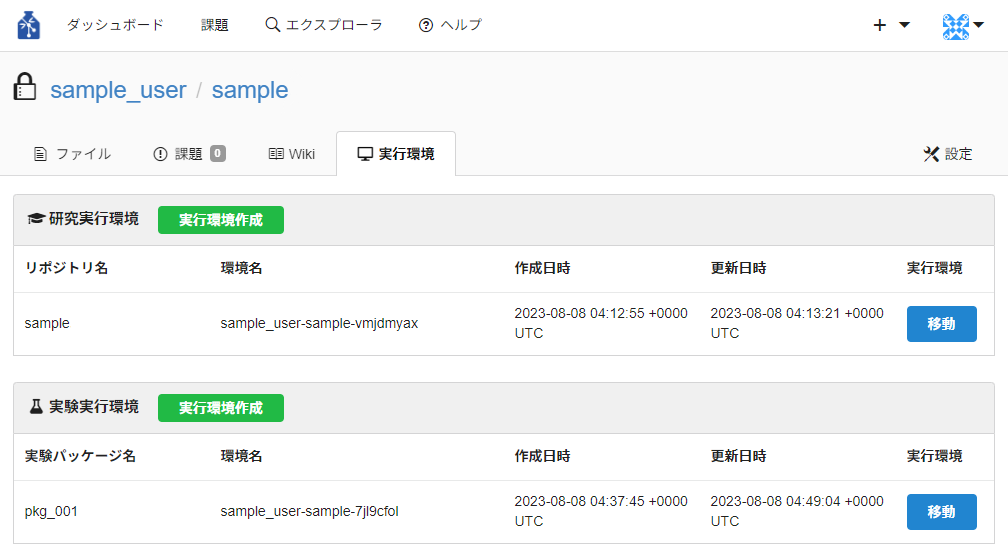
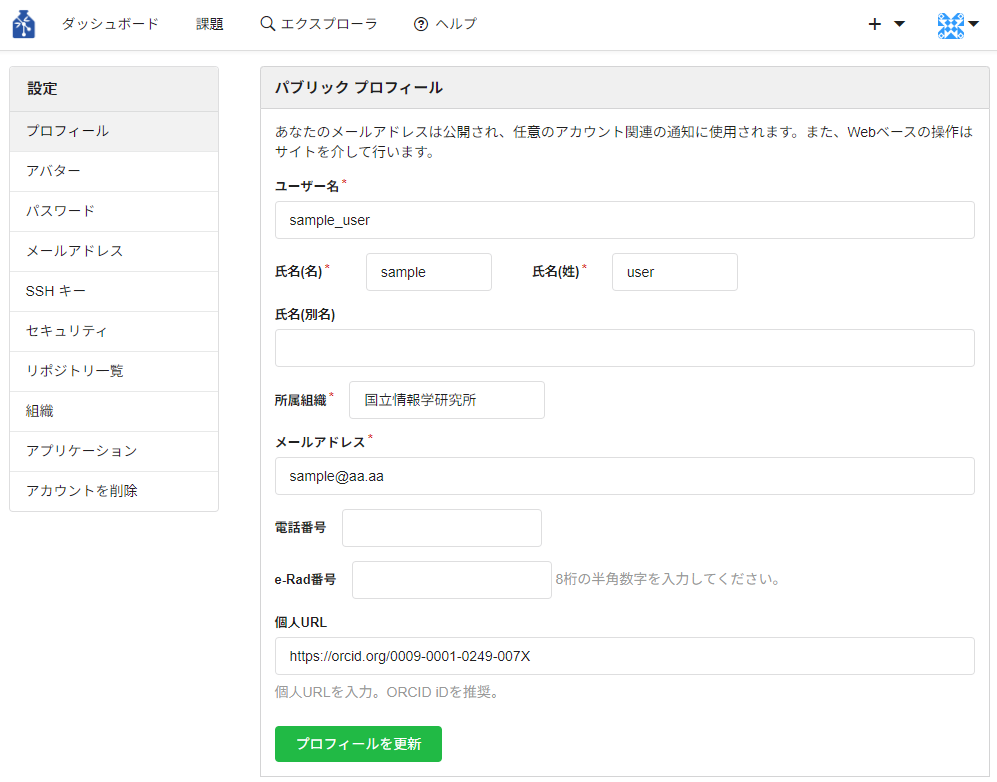
- ユーザー設定画面
- プロフィール
自分のユーザー情報を変更することができます。
- セキュリティ
2段階認証の有効/無効を設定することができます。初期設定では状態が無効を意味する「OFF ×」となっています。
※ 使用する場合は一般公開されている認証アプリが必要です。PCもしくは携帯端末に予めダウンロードをお願いします。
※ 動作確認済み認証アプリ:Google Authenticator、Microsoft Authenticator、IIJ SmartKey、WinAuth、Step Two
※ 以下は携帯端末にて設定する方法の説明になります。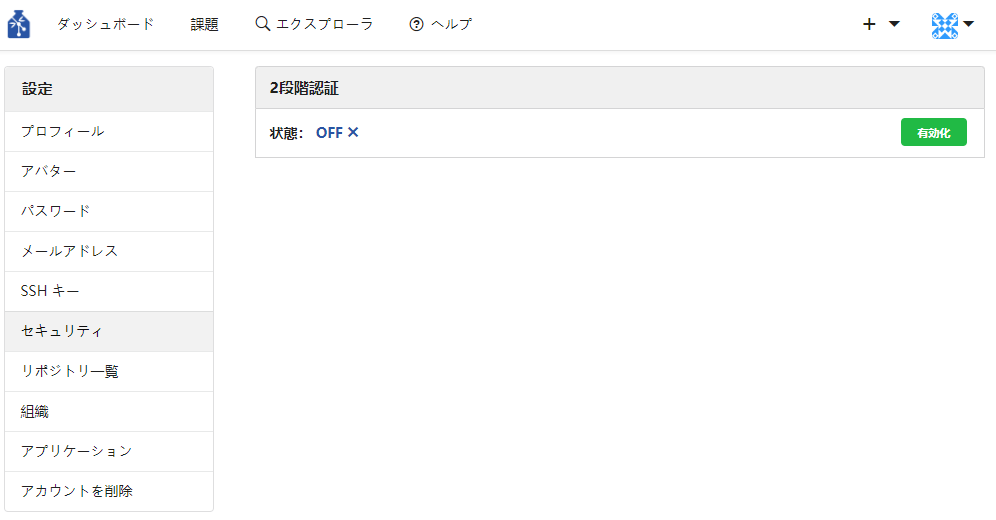
「有効化」のボタンを押下すると、2次元コードが表示されます。認証アプリで読み込むかシークレットコードを入力してください。
認証アプリ側に「GIN-fork:ユーザーのメールアドレス」のエリアが出来、6桁の数字が表示されます。
「パスコードを入力してください:」のボックスに、表示された6桁の数字を入力してください。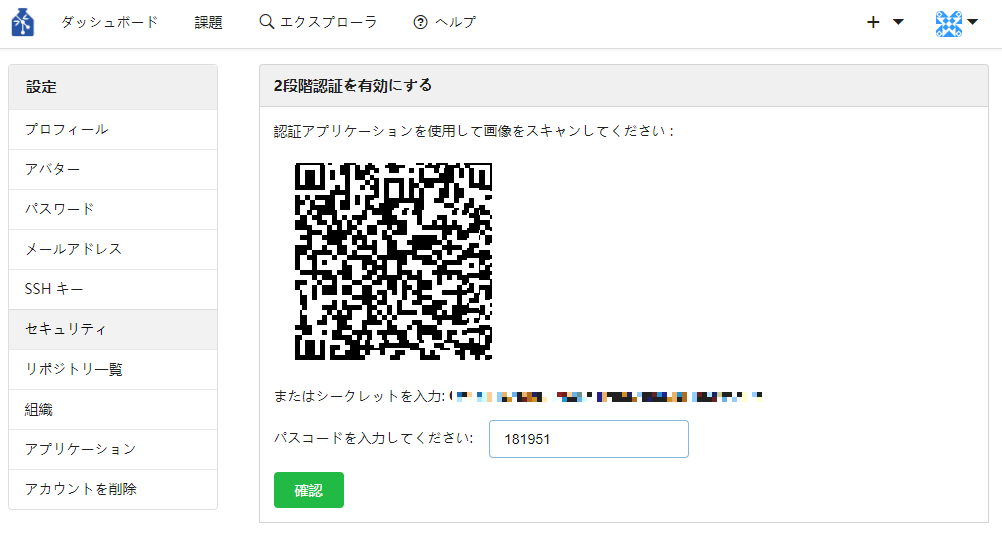
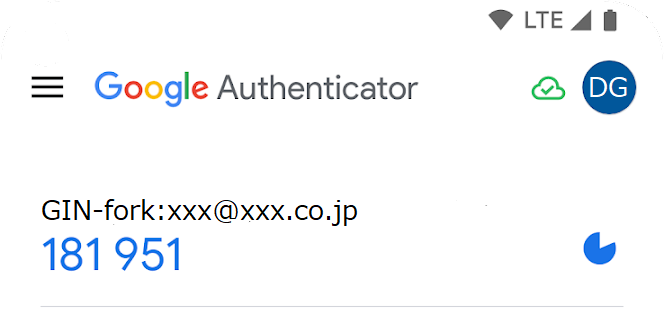
成功すると、有効化された旨のメッセージとリカバリーコードが表示されます。
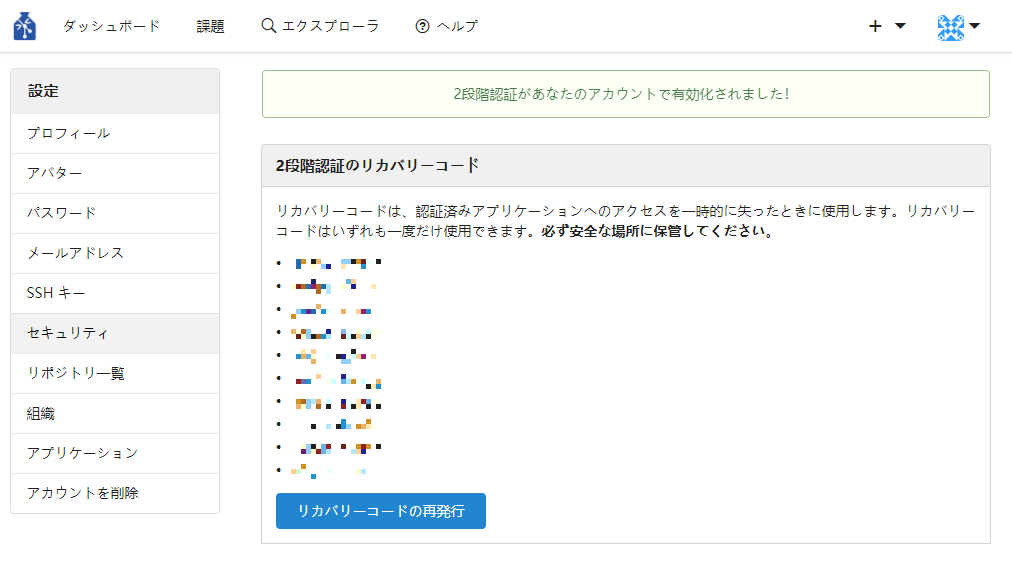
再度セキュリティ設定画面に遷移すると、状態が有効を意味する「ON ✓」となります。無効に戻したい場合は「無効にする」のボタンを押下してください。
※ 無効にした場合は認証アプリ側も削除してください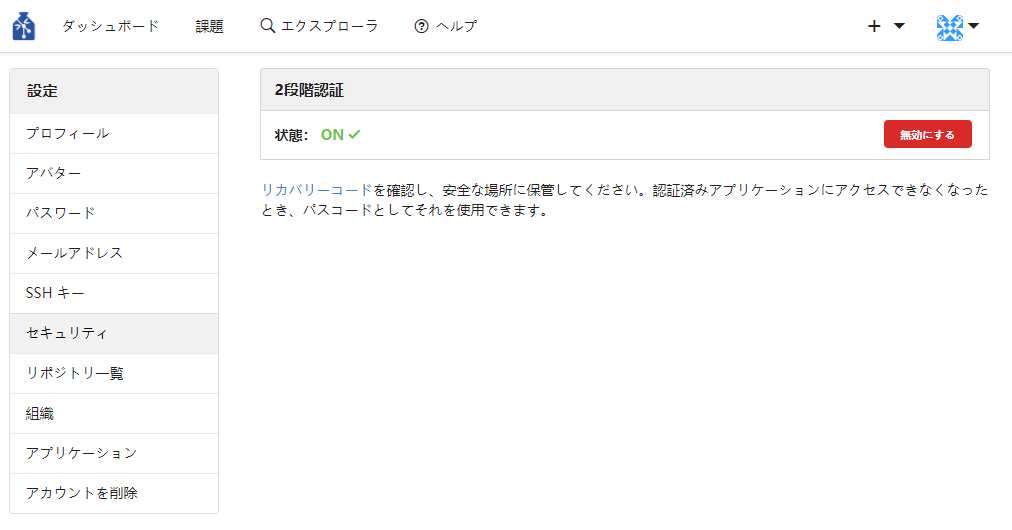
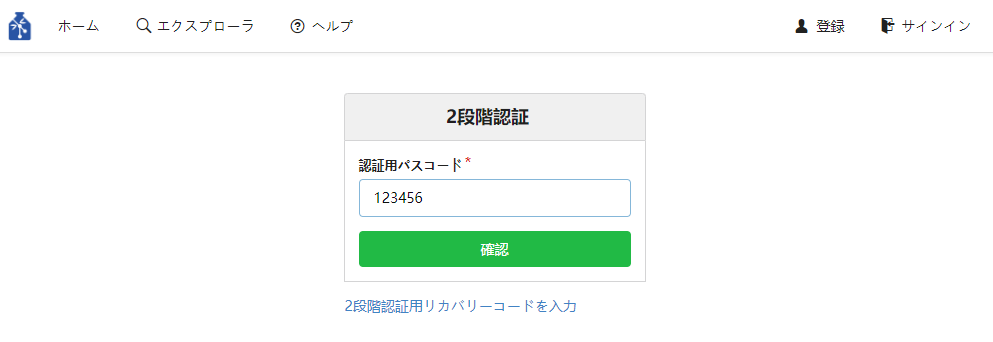
2段階認証を行うと、サインイン時にコード入力画面が表示されます。認証アプリを起動して6桁の数字を「認証用パスコード」の欄に入力してください。
- プロフィール
- リポジトリ設定画面
- オプション
リポジトリに関する設定を行うことができます。
リポジトリを公開にする場合は「このリポジトリは非公開です」のチェックを外してください。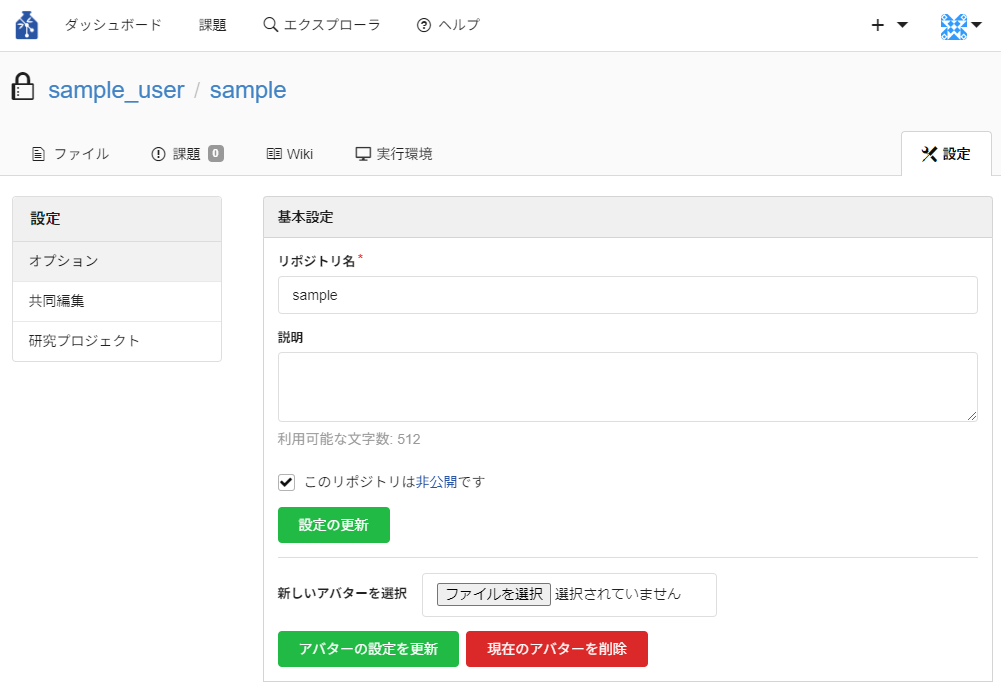
- 共同編集
リポジトリに共同編集者とするユーザーを追加することができます。
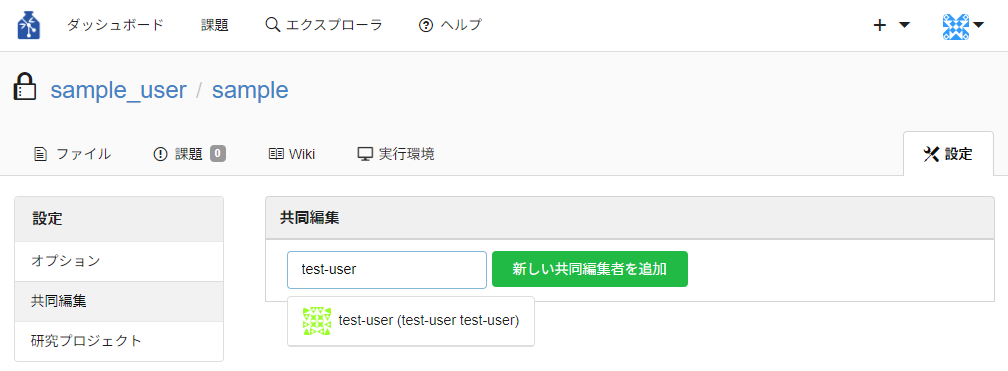
共同編集者のリポジトリに対する権限はデフォルトでは書込となっています。この権限を管理・書込・読込に変更することができます。
各権限の操作範囲は『9.リポジトリに対する権限レベル』を参照してください。
共同編集者を削除する場合は「削除」ボタンを押下して削除してください。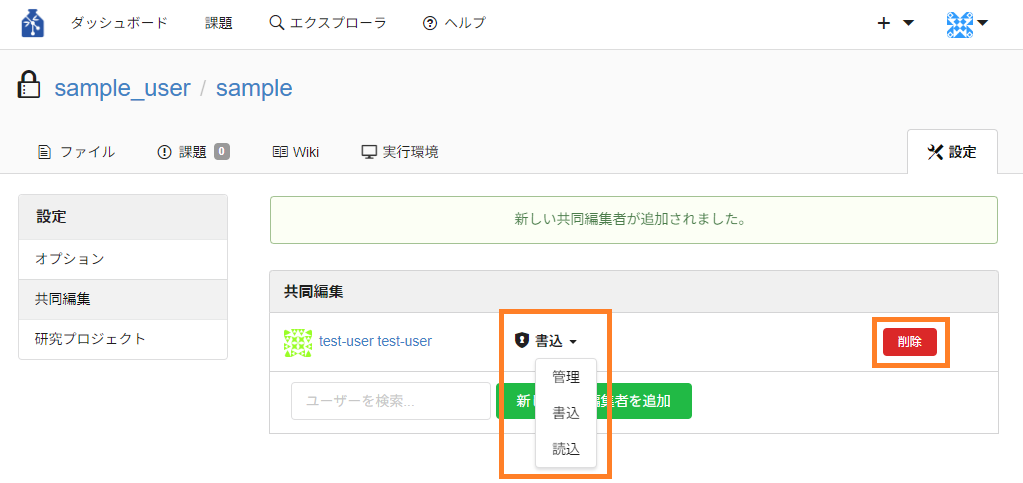
- 研究プロジェクト
研究プロジェクト名と研究プロジェクトの説明の設定を行うことができます。
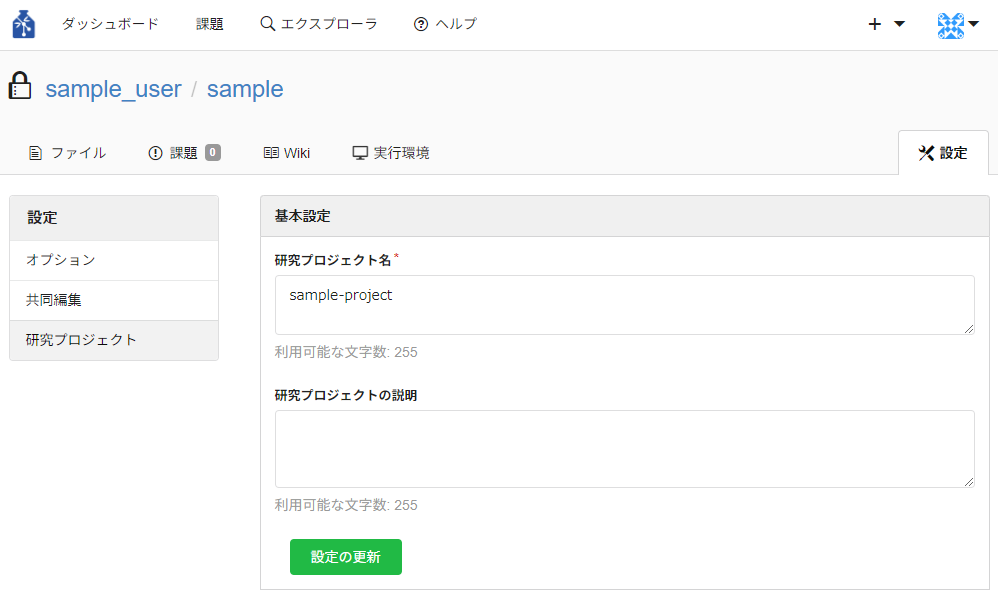
- オプション
- ファイルコミット
ファイルの作成・削除・更新時には、メッセージと説明をつけることができます。
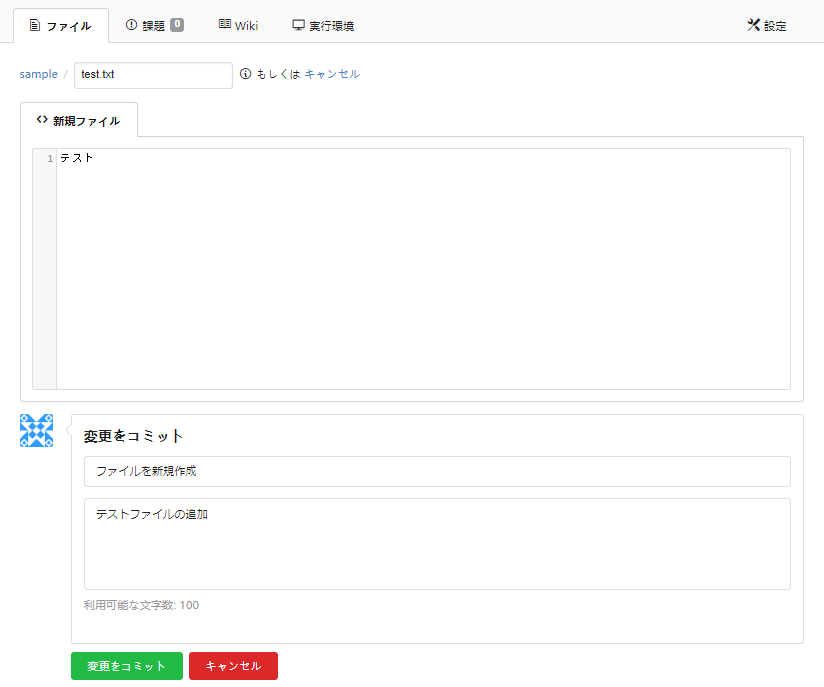
これらはファイルの変更差分とともに確認することができます。
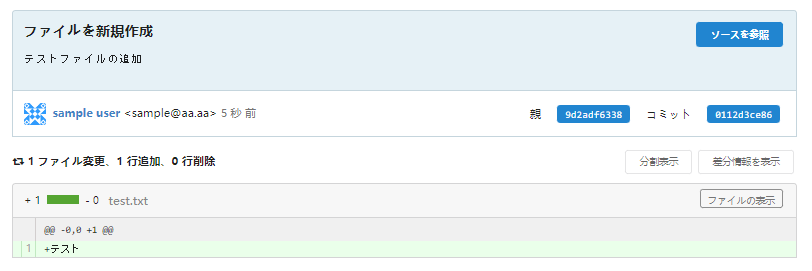
上記の画面は、コミット履歴やリポジトリトップ画面等に表示されるハッシュ値(SHA1)をクリックすると閲覧できます。
- リポジトリに対する権限レベル
リポジトリに対する操作権限は「オーナー」、「管理」、「書込」、「読込」となります。
各権限にて操作できる内容は以下となります。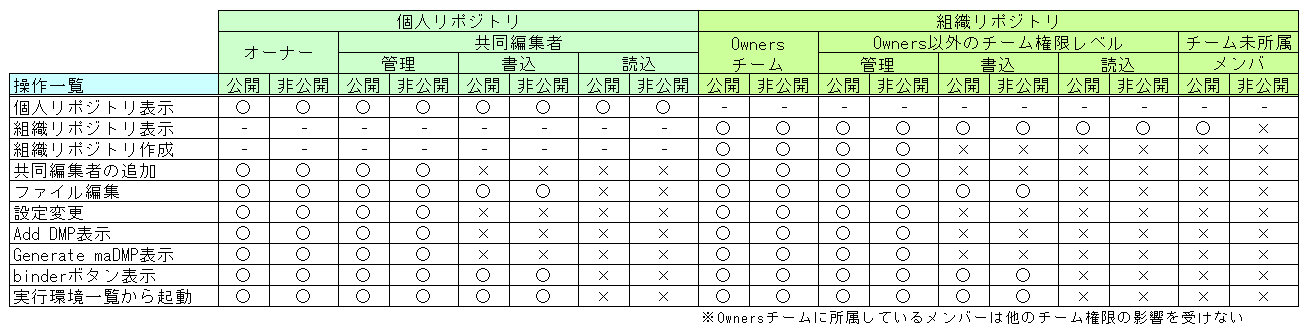
- 組織とリポジトリ
組織を作成すると、組織のリポジトリを作成することができます。
組織のリポジトリは所属するメンバーで研究・実験を行うことができます。
組織の作成やメンバー追加の手順については以下の手順で実施してください。- 組織を作成する
① ページ右上の「+」(作成)をクリックしドロップダウンメニューで「新しい組織」をクリック、またはダッシュボードの左上にあるユーザー名をクリックし「+ 新しい組織」をクリック、または組織タブの「+」(新しい組織)をクリックします。
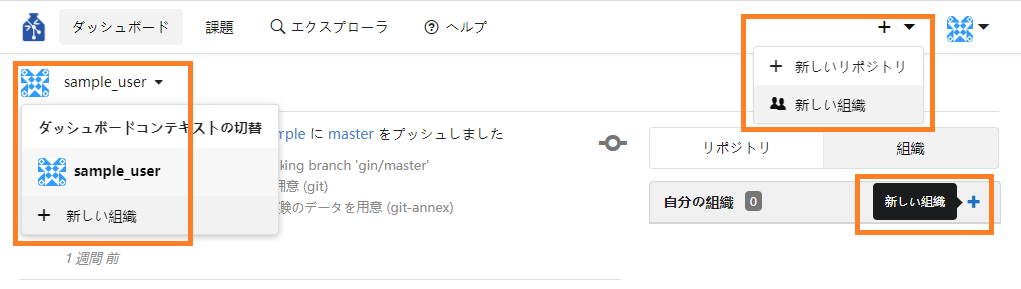
② 組織名を入力するページに遷移します。組織名を入力して「組織の作成」ボタンを押下してください。
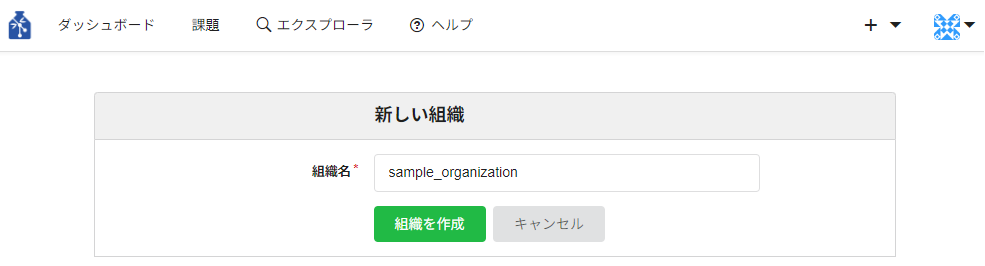
③ ボタン押下後、組織のダッシュボードに遷移します。右上の「"組織名"を見る」をクリックすると、組織のページに遷移します。
組織名の変更などを行いたい場合は、組織ページの組織名の右の「⚙」をクリックすると、変更画面に遷移します。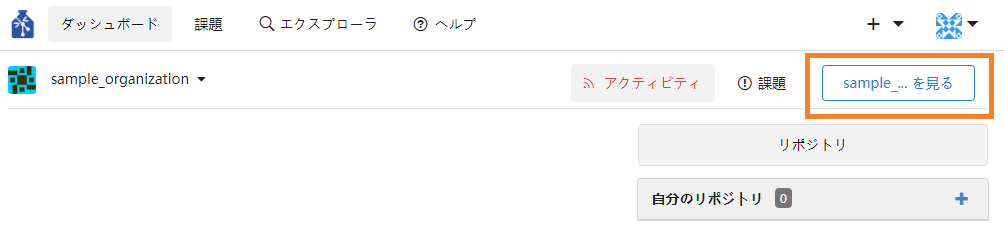
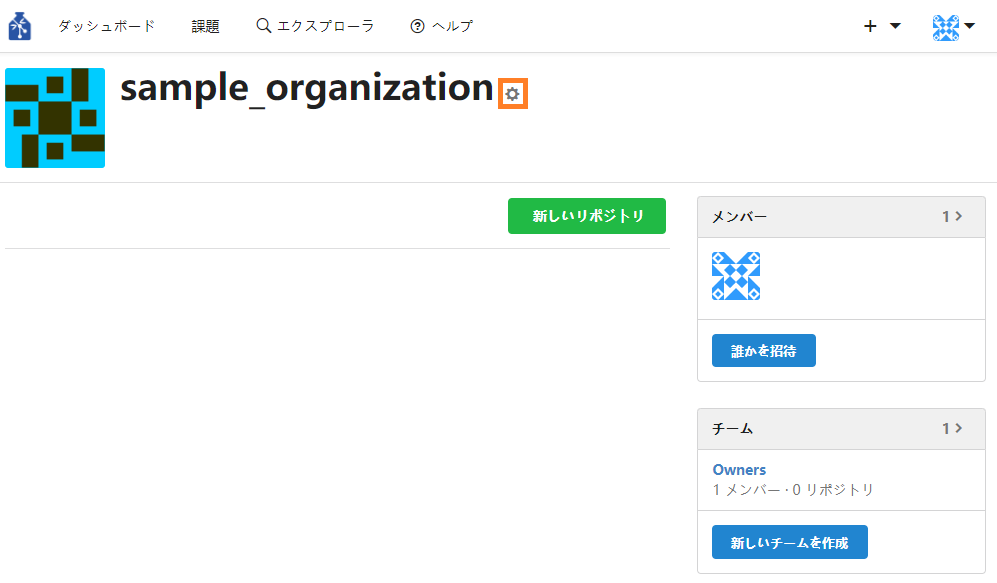
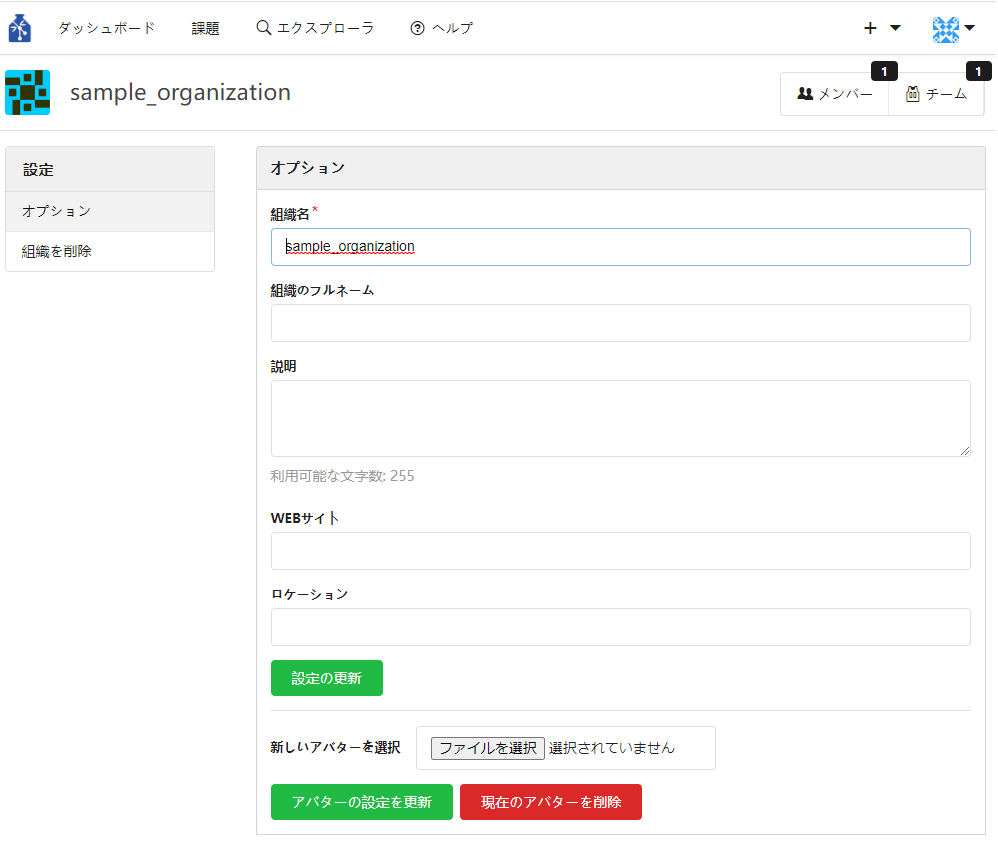
④ 組織を作成後、自分のダッシュボードから組織のページに遷移する場合は以下の操作で遷移することができます。
・ページ左上のユーザー名をクリックし、ドロップダウンメニューから組織を選択すると、組織のダッシュボードに遷移します。右上の「"組織名"を見る」をクリックすると、組織のページに遷移します。
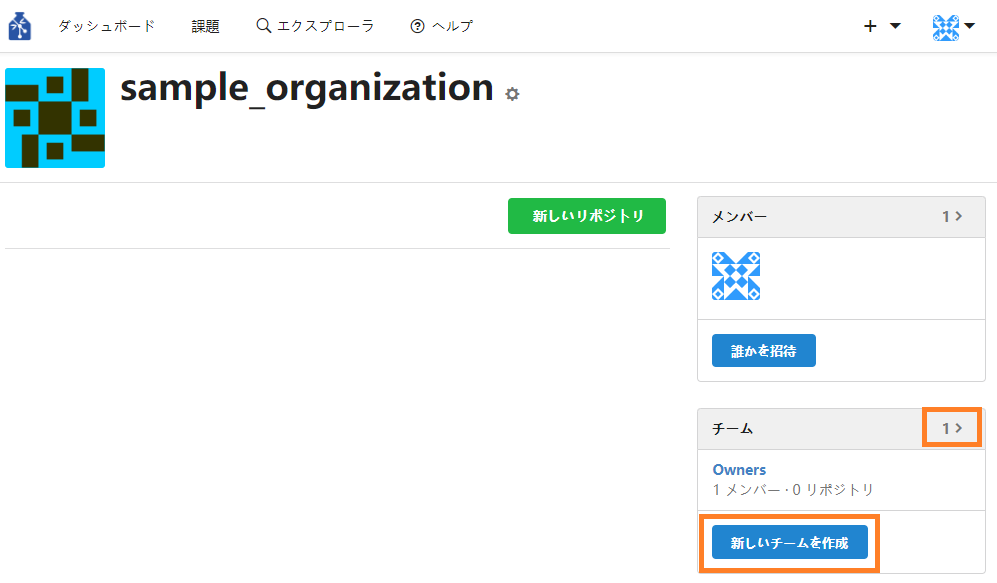
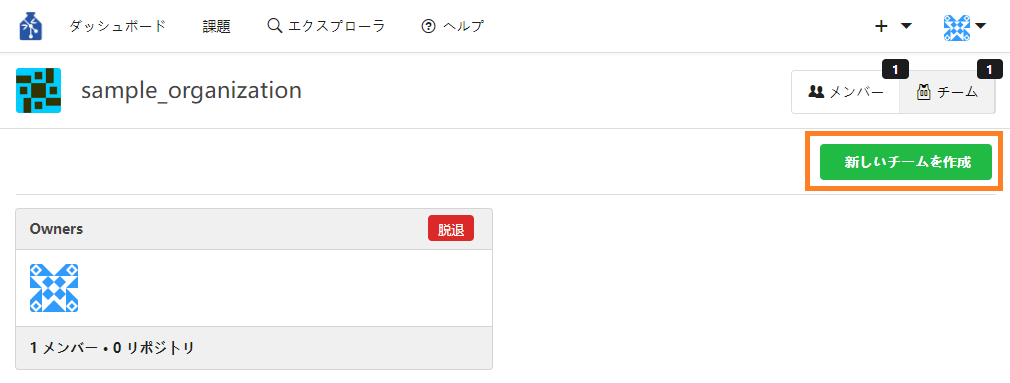
・右側の「組織」のタブをクリックし組織を選択すると、組織のページに遷移します。 - 組織内でチームを作成する
組織を作成すると、組織のオーナーで構成されているチーム「Owners」がデフォルトで作成されます。それ以外のチームを作成する場合は以下の方法で作成してください。
「Owners」については『7.組織メンバーを組織のオーナーにする』を参照してください。① 組織ページの右下の「新しいチームを作成」のボタンを押下、または「チーム」エリア右の「"数字" >」をクリックしてチームページに遷移後、「新しいチームを作成」ボタンを押下します。
② チーム名と簡単な説明を入力します。チームの権限レベル (読み取り、書き込み、管理者) を選択し、「新しいチームの作成」のボタンを押下します。
押下後チームのページに遷移します。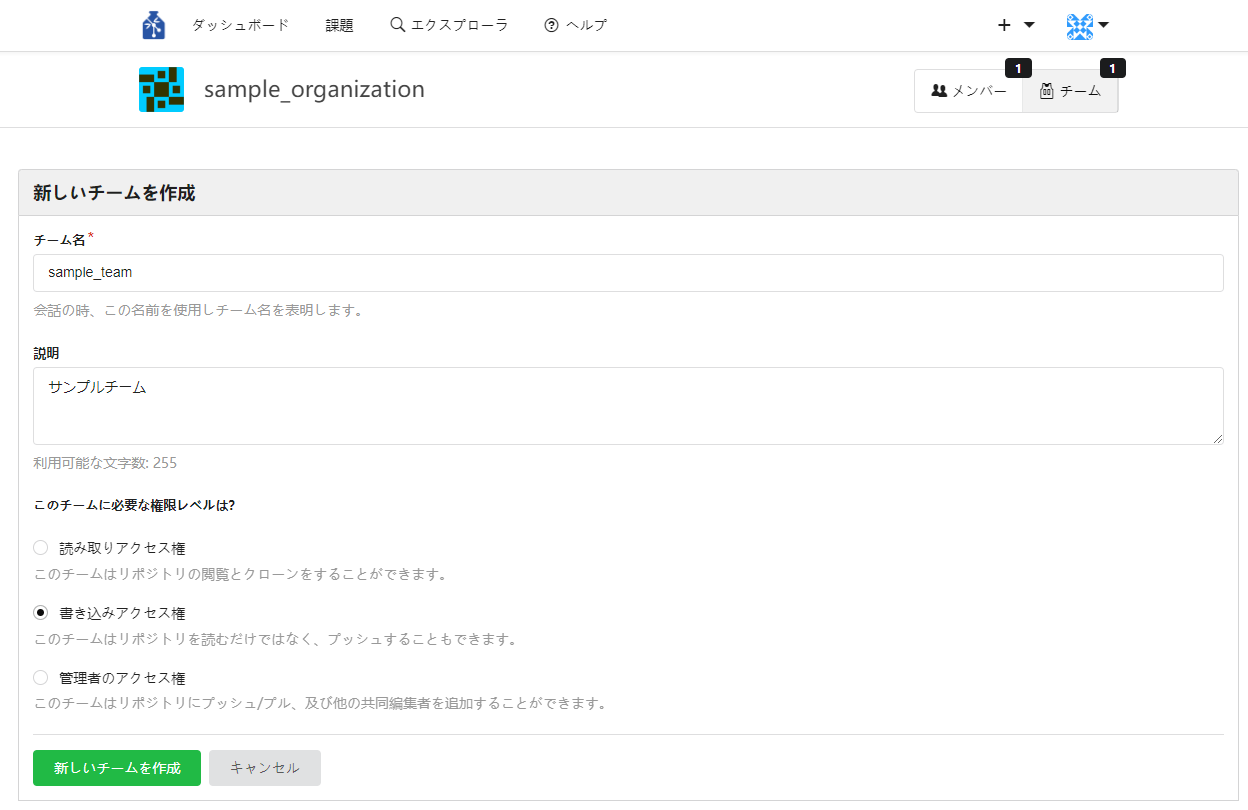
③ チーム名や権限について設定を変更したい場合はチームのページの「設定」ボタンを押下すると、設定画面に遷移します。
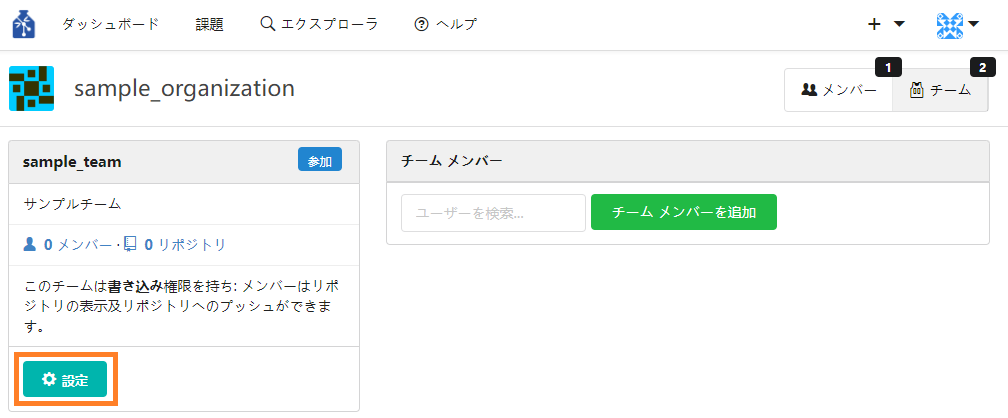
- チームにメンバーを追加する
① 組織のページの「チーム」エリアからチーム名をクリックし、チームのページに遷移します。
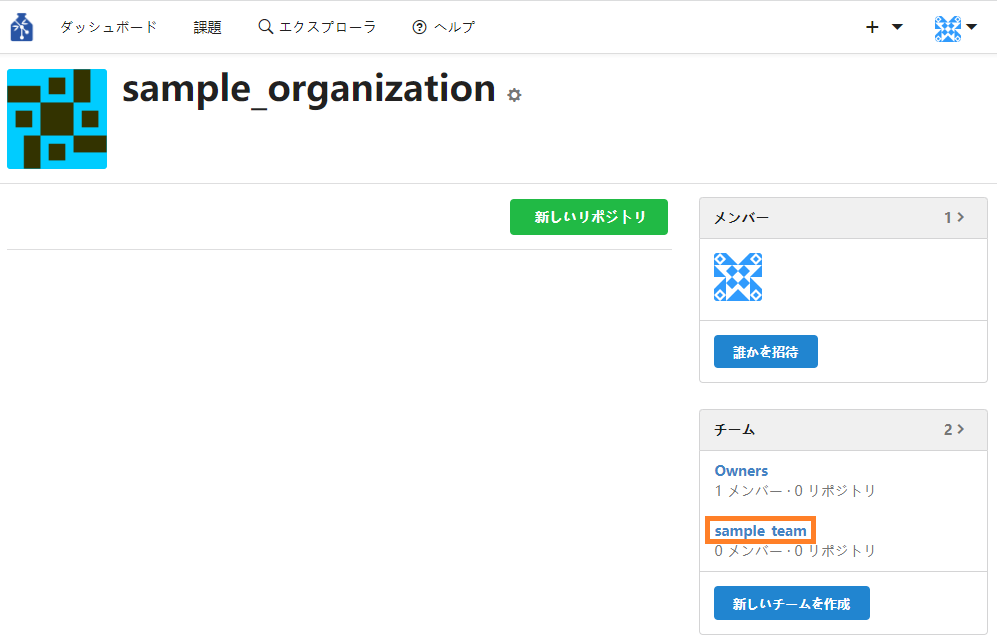
② チームのページの「チームメンバー」のエリアの検索ボックスで追加したいメンバーを検索し指名します。指名後「チームメンバーを追加する」ボタンを押下します。
押下後、指名したメンバーがチームメンバーとなります。自身がオーナーの場合はチーム名右の「参加」ボタンを押下することでチームメンバーになることもできます。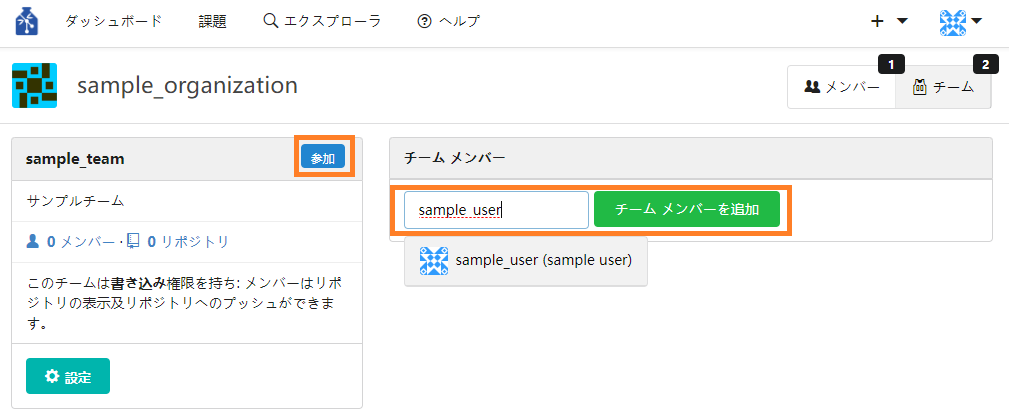
③ チームメンバーを削除したい場合は、メンバー名の右側の「削除」ボタンを押下してください。自身がオーナーの場合はチーム名右の「脱退」ボタンを押下することでチームメンバーから脱退することができます。
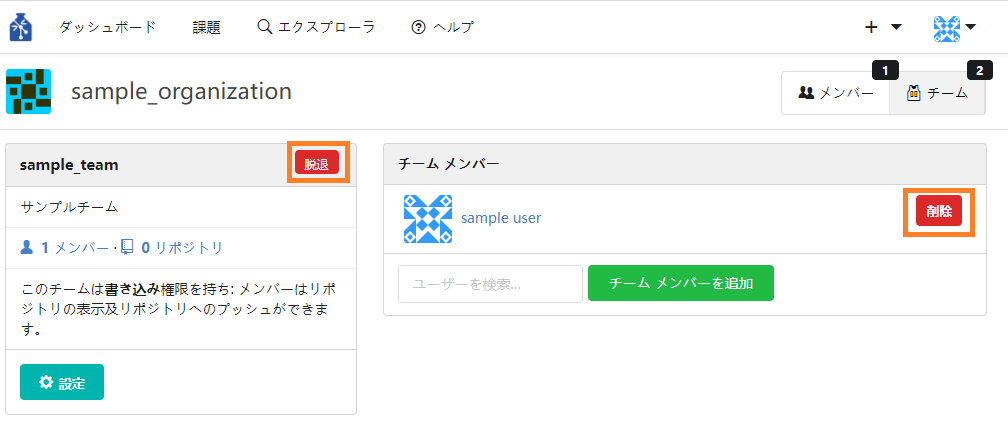
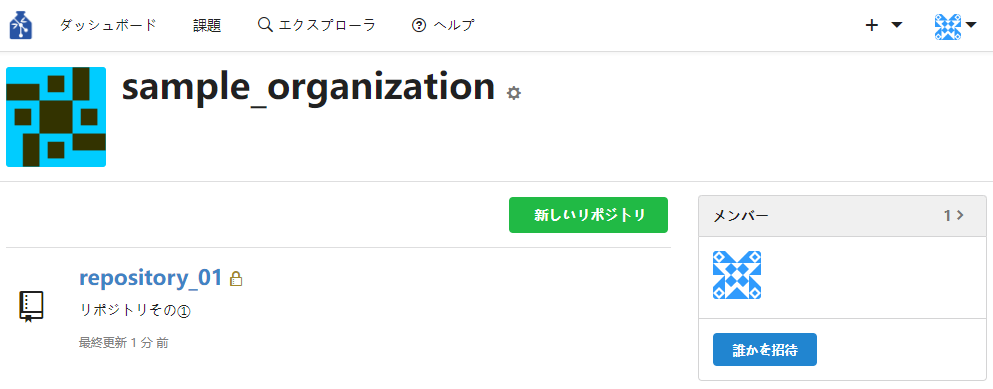
- 組織へのリポジトリを追加する
その組織のオーナーのみが組織にリポジトリを追加することができます。オーナーは組織ページのチーム「Owners」に所属するメンバーとなります。
「Owners」については『7.組織メンバーを組織のオーナーにする』を参照してください。① 組織ページの「新しいリポジトリ」のボタンを押下します。または組織のダッシュボードの「リポジトリ」エリアの「+」(新しいリポジトリ)をクリックします。
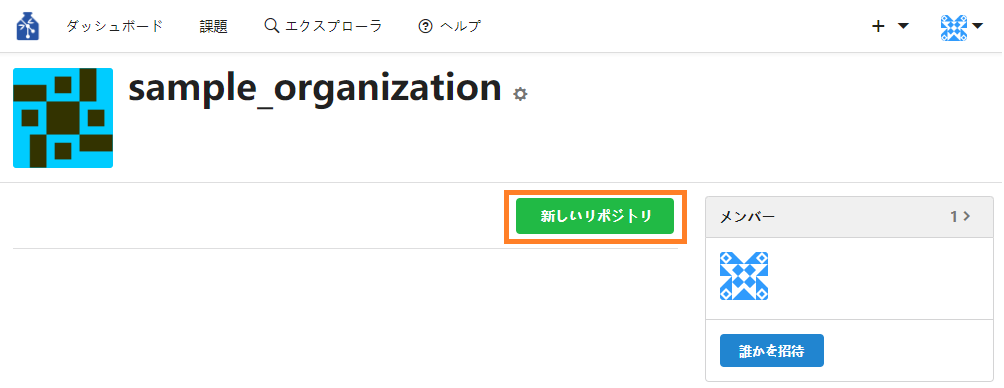
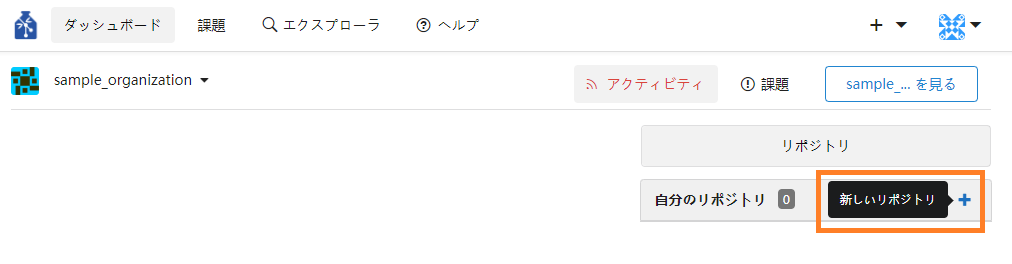
② オーナーがリポジトリ作成対象の組織になっていることを確認します。必要事項を入力しページの下部にある「リポジトリの作成」ボタンを押下します。
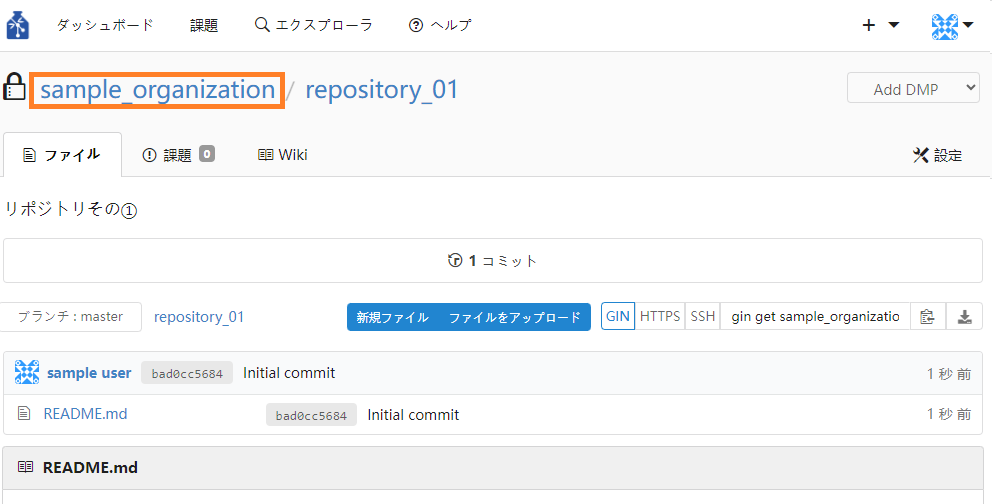
押下後、作成したリポジトリに遷移します。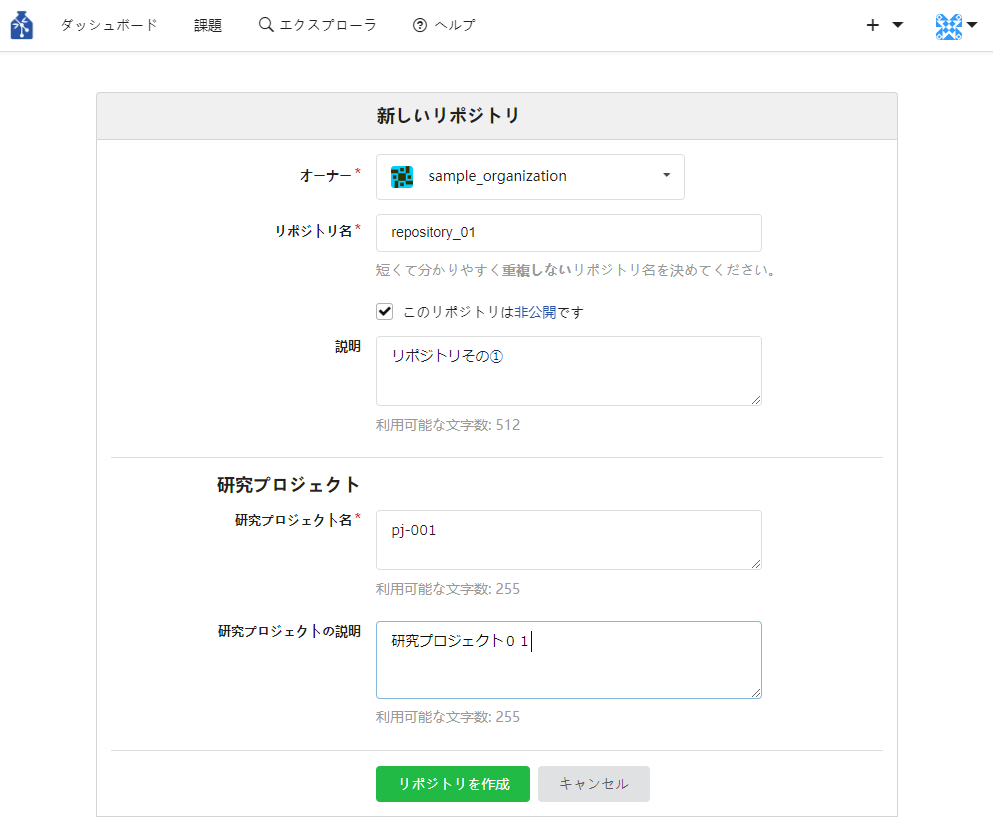
③ リポジトリ画面上部の組織名をクリックすると、組織ページに戻ることができます。
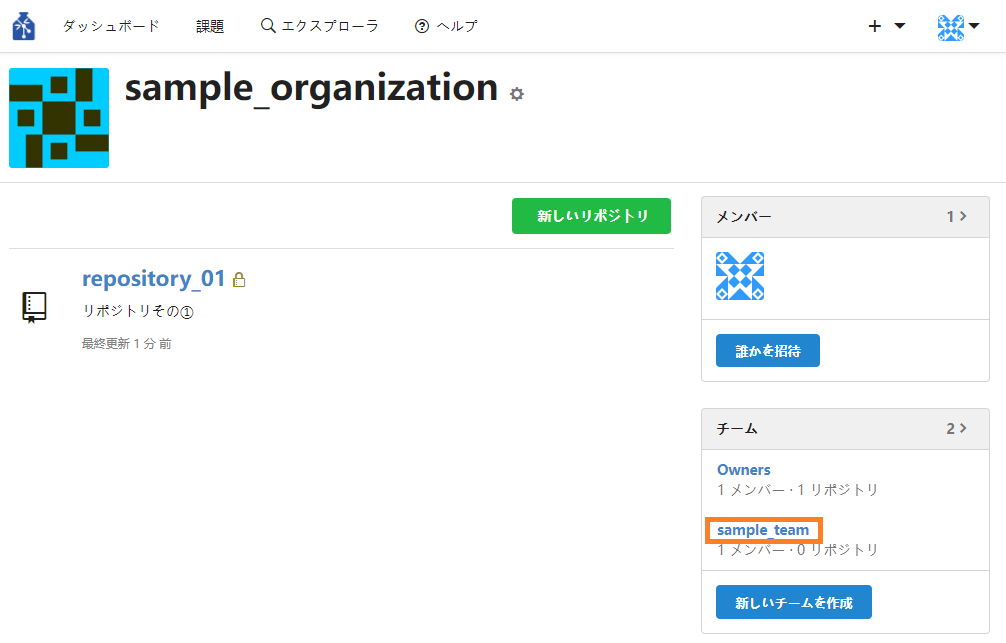
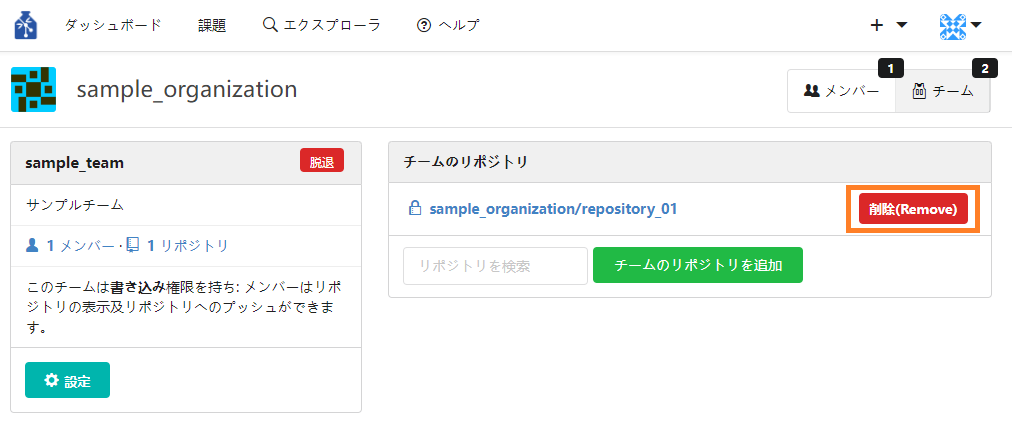
- チームにリポジトリを追加する
※ リポジトリに対するメンバーの操作権限は、チーム作成時に設定したチームの権限レベルとなります。
① 組織ページの右下にある「チーム」リストからチーム名をクリックし、チームのページに遷移します。
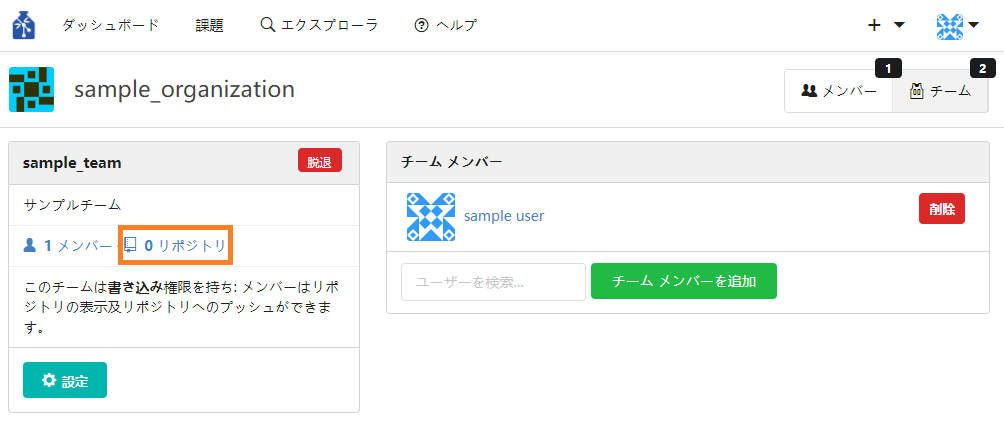
② ページ左側の「メンバー」リンクの隣にある「リポジトリ」のリンクをクリックします。
③ ページ右側にチームのリポジトリが表示されます。検索ボックスでリポジトリを検索し指定します。指定後「チームのリポジトリを追加」ボタンを押下します。
押下後、指定したリポジトリがチームのリポジトリとなります。
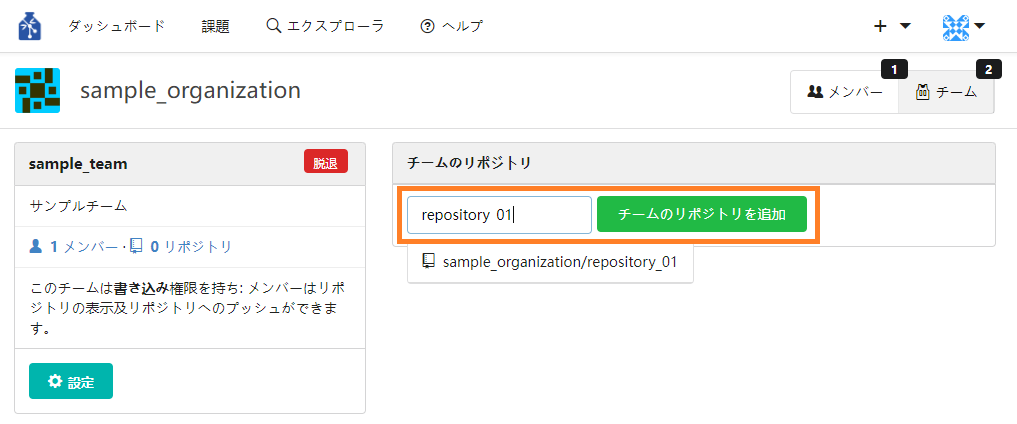
④ リポジトリをチームから削除したい場合は、右側の「削除(Remove)」ボタンを押下してください。
- 組織を作成する
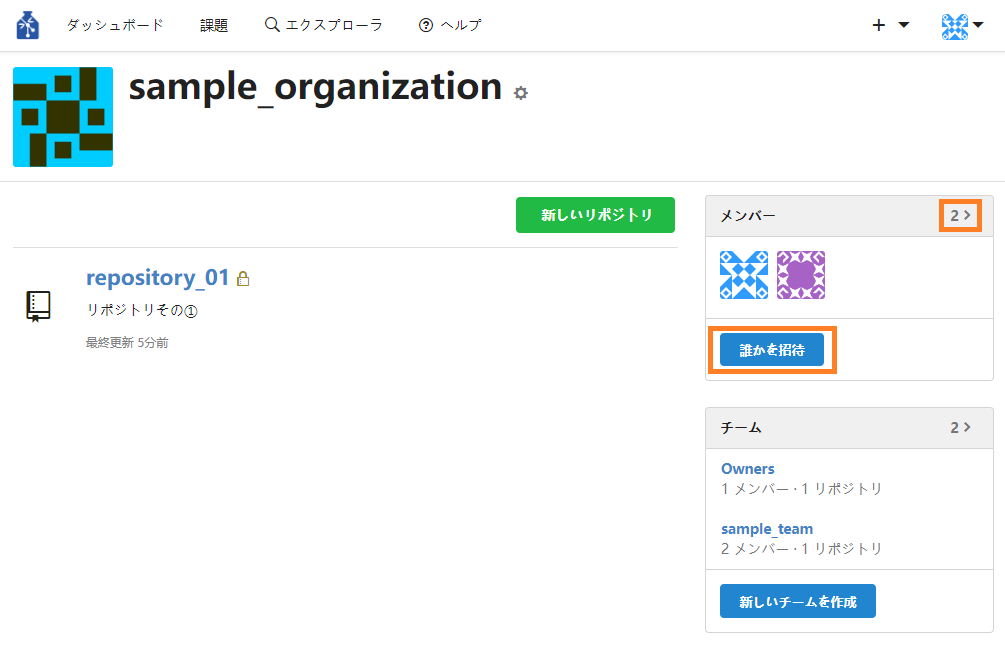
- 組織にメンバーを追加する
組織のメンバーはチームにメンバーを追加した際に自動で追加されますが、チームに所属せずにメンバーのみ追加することもできます。
① 組織ページの右側にある「メンバー」エリアの「誰かを招待」ボタンを押下、または「メンバー」エリア右の「"数字" >」をクリックしてメンバーページに遷移後、「誰かを招待」ボタンを押下します。
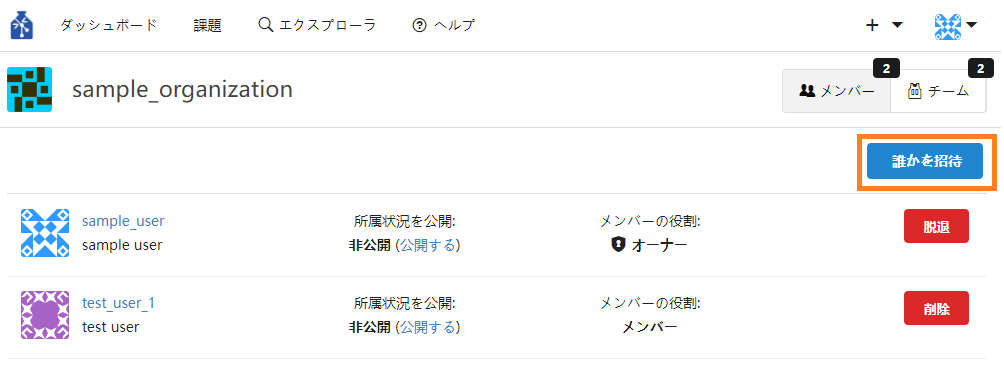
② 検索ボックスで追加したいメンバーを検索し指名します。指名後「今すぐ招待」ボタンを押下します。
押下後、指名したメンバーが組織のメンバーとなります。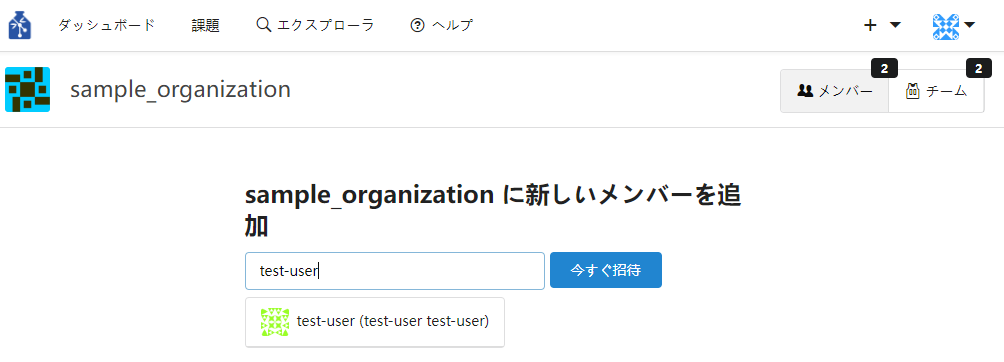
③ 組織からメンバーを削除したい場合は、メンバー名の右側の「削除」ボタンを押下してください。
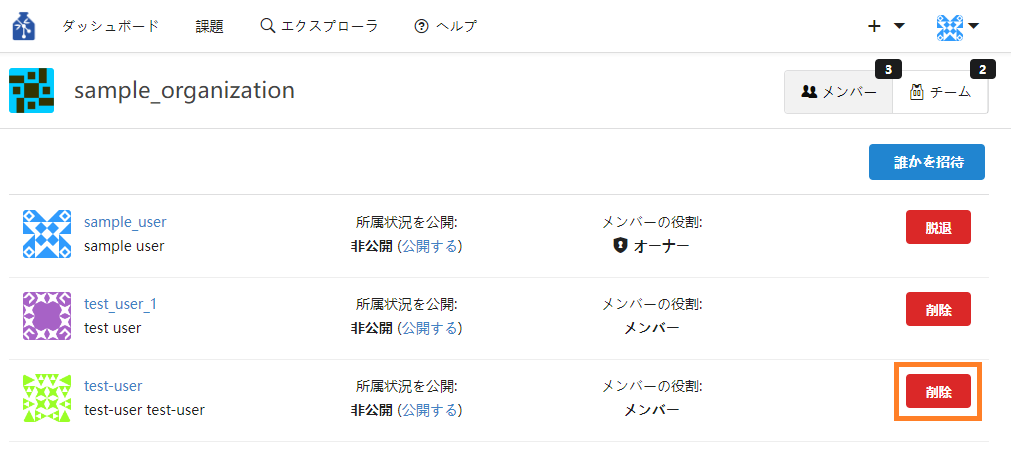
- 組織メンバーを組織のオーナーにする
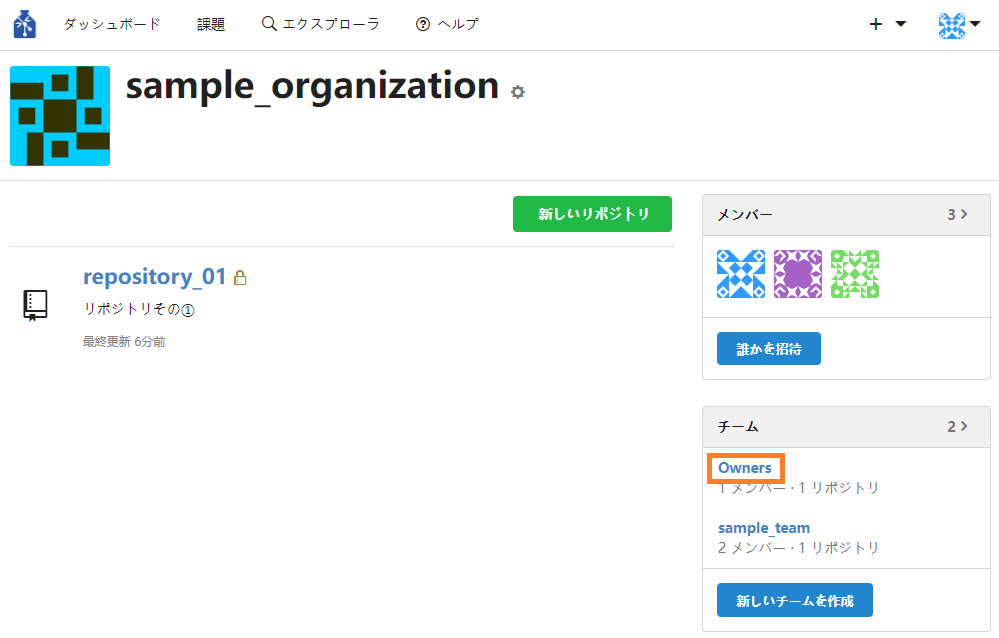
組織内のメンバーに組織のオーナー権限を付与するには、オーナーがメンバーを「Owners」に追加する必要があります。
① 組織ページの右下にある「チーム」エリアから「Owners」をクリックしてオーナーチームのページに遷移します。
② 「チームメンバー」のエリアの検索ボックスで追加したいメンバーを検索し指名します。指名後「チームメンバーを追加する」ボタンを押下します。
押下後、指名したメンバーが組織のオーナーとなります。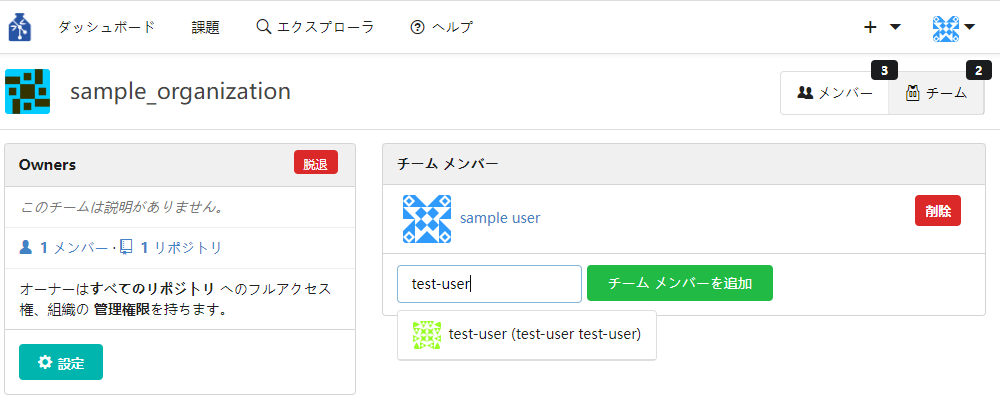
③ オーナー権限を削除したい場合は、メンバー名の右側の「削除」ボタンを押下してください。
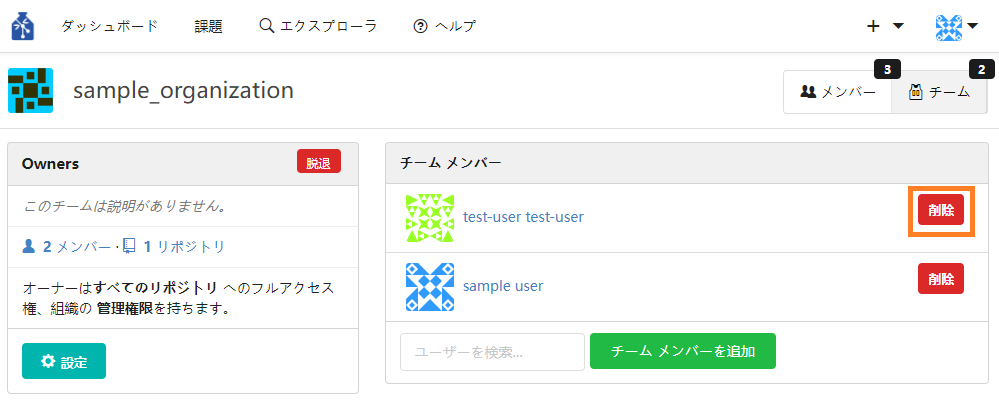
④ オーナー権限を持つメンバーはメンバーページにて、メンバーの役割が「オーナー」となってることが確認できます。
付録2 Jupyter Notebookについて
PythonなどをWebブラウザ上で記述・実行できる対話型の統合開発環境です。
もともとはIPython NotebookというPython専用環境でしたが、Jupyter NotebookではJulia、Rなど40以上の言語がサポートされています。
Jupyter Notebook上では、様々なライブラリを使うことができ、機械学習やディープラーニング用のライブラリも動作させることができます。
※ 現段階のデータガバナンス機能ではPythonでのみ実験を行うことができます。
Jupyter Notebook操作方法
- Jupyter Notebookの操作について
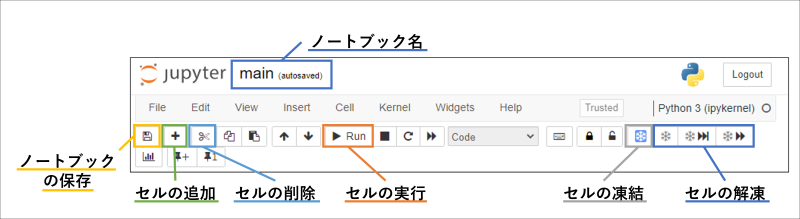
- 操作ボタン
Jupyter notebookでは、画面上部のボタンを使用してノートブックやセルを操作することができます。
また、「Help」の「keyboard Shortcuts」からショートカットキーを確認できます。
- 折り畳み表示
セルが折り畳まれ、コードが非表示になっている個所は、
 ボタンにてセルをまとめて実行することができます。
ボタンにてセルをまとめて実行することができます。
▷をクリックすると、折り畳まれて非表示になっているセルを開くことができます。
再度▽をクリックすると、折り畳まれた状態に戻ります。
- ステータス確認
実行完了、凍結、解凍などのセルの状態は視覚的に確認することができます。
以下の図は、セルの表示例です。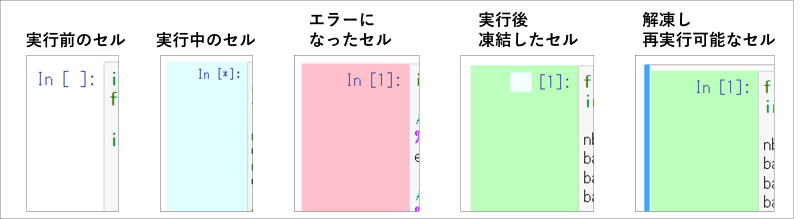
また、各セルの実行結果は以下のように、ステータス毎に異なる背景色で出力されます。
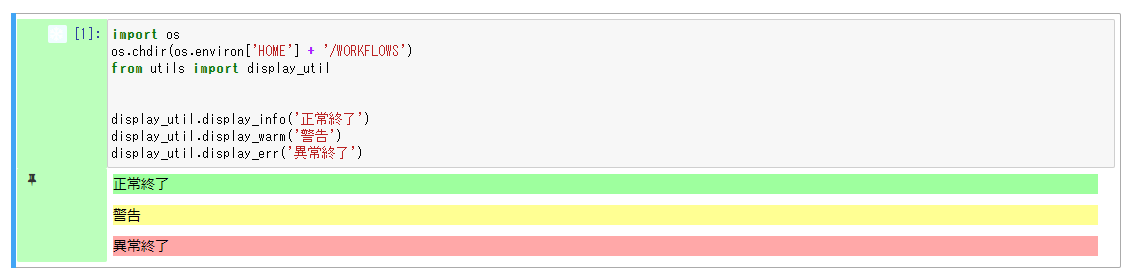
- 操作ボタン
- セルを実行する
セルを実行する際は、画面上部の
 ボタン、またはセル内の
ボタン、またはセル内の ボタンをクリックしてください。
ボタンをクリックしてください。データガバナンス機能が提供する環境では、実行したセルは凍結し、そのままではセルの編集や再実行ができなくなっています。
一度実行したセルを再実行する場合はセルを解凍してください。■ 注意事項
ツールバーのCellメニューより選択し実行することも可能ですが、タスクNotebookを実行すると入力(例:ユーザー名、パスワードなど)を求められる場合があります。
入力項目を入力せずに「Run All」や「Run All Below」を実行すると、未入力のまま後続のセルが実行されるためエラーとなります。
エラーとなった場合は、入力項目のセルより再実行をしてください。 - セルを解凍する
データガバナンス機能が提供する環境では、国立情報学研究所が開発した拡張機能により、一度実行したセルは凍結され、解凍しない限りセルの編集や再実行ができなくなっています。
拡張機能についてはこちらをご覧ください。一度実行したセルを編集・再実行したい場合は、以下の説明のようにセルの解凍を行ってください。
- 選択したセルを解凍する
 をクリックすると、選択したセルを解凍することができます。
をクリックすると、選択したセルを解凍することができます。 - 同セクション内のセルを解凍する
 をクリックすると、選択したセル以降の同じセクション内にあるセルを解凍することができます。
をクリックすると、選択したセル以降の同じセクション内にあるセルを解凍することができます。 - 全てのセルを解凍する
 をクリックすると、選択したセル以降の全てのセルを解凍することができます。
をクリックすると、選択したセル以降の全てのセルを解凍することができます。
- 選択したセルを解凍する
- セルを追加する
セルを追加する際は、任意の位置を選択した後に、画面上部の「+」ボタンを押してください。
- セルを削除する
追加したセルを削除したい場合は、削除したいセルを選択し、画面上部のハサミの描かれたボタンをクリックしてください。
- システムコマンドを利用する
Jupyter notebookでは、!を先頭につけることでシステムコマンド(シェルコマンド)を実行することができます。
- Jupyter Notebookのその他の操作について
詳細はUser Documentationをご確認ください。
付録3 その他機能について
リサーチフロー:共通メニュー
タスクNotebookではフェーズやタスクに関わらず共通な項目を共通メニュー化しています。
共通メニューの各項目は以下となります。
- 研究リポジトリ名を確認する
主に研究フロー図の各タスクのタスクNotebookにて表示される項目です。
研究実行環境で操作する研究リポジトリ名を確認できます。
研究リポジトリ名を変更した際は再選択し、再確認してください。
※ 非公開リポジトリでは、初期セットアップ実施前にリポジトリ名を変更した場合に最新のリポジトリ名が表示されません。
初期セットアップ実施後に再選択して確認してください。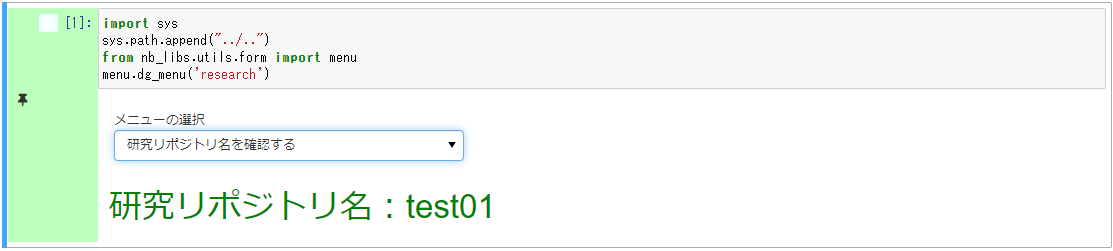
- 研究リポジトリ名・実験パッケージ名を確認する
主に実験フロー図の各タスクのタスクNotebookにて表示される項目です。
実験実行環境で操作する研究リポジトリ名と研究リポジトリの実験パッケージの名前を確認できます。
表示された名前の実験パッケージ内の変更がGIN-forkへ同期されます。
研究リポジトリ名を変更した際は再選択し、再確認してください。
※ 非公開リポジトリでは、初期セットアップ実施前にリポジトリ名を変更した場合に最新のリポジトリ名が表示されません。
初期セットアップ実施後に再選択して確認してください。
※ 実験パッケージ名は、初期セットアップ実施前など未設定の場合ハイフンで表示されます。
初期セットアップで実験パッケージ名を決定した後に再選択すると、設定した実験パッケージ名が確認できます。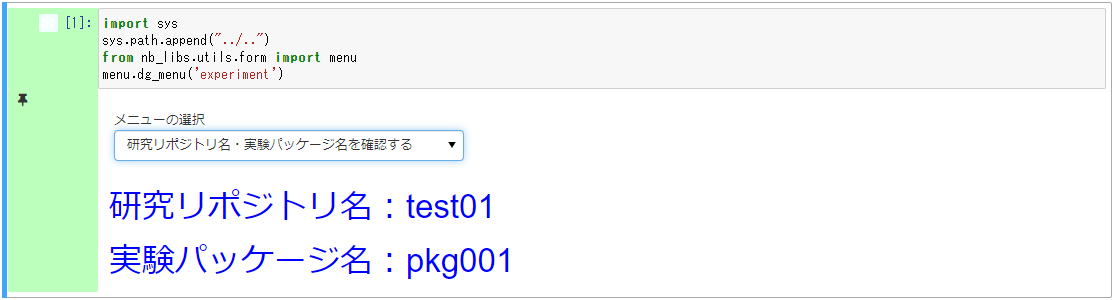
- 研究フロートップページに遷移する
主に研究フロー図の各タスクのタスクNotebookにて表示される項目です。
研究フロートップページへ遷移するボタンを表示します。
- 実験フロートップページに遷移する
主に実験フロー図の各タスクのタスクNotebookにて表示される項目です。
実験フロートップページへ遷移するボタンを表示します。
- GIN-forkに遷移する
すべての共通メニューにて表示される項目です。
GIN-forkへ遷移するボタンを表示します。
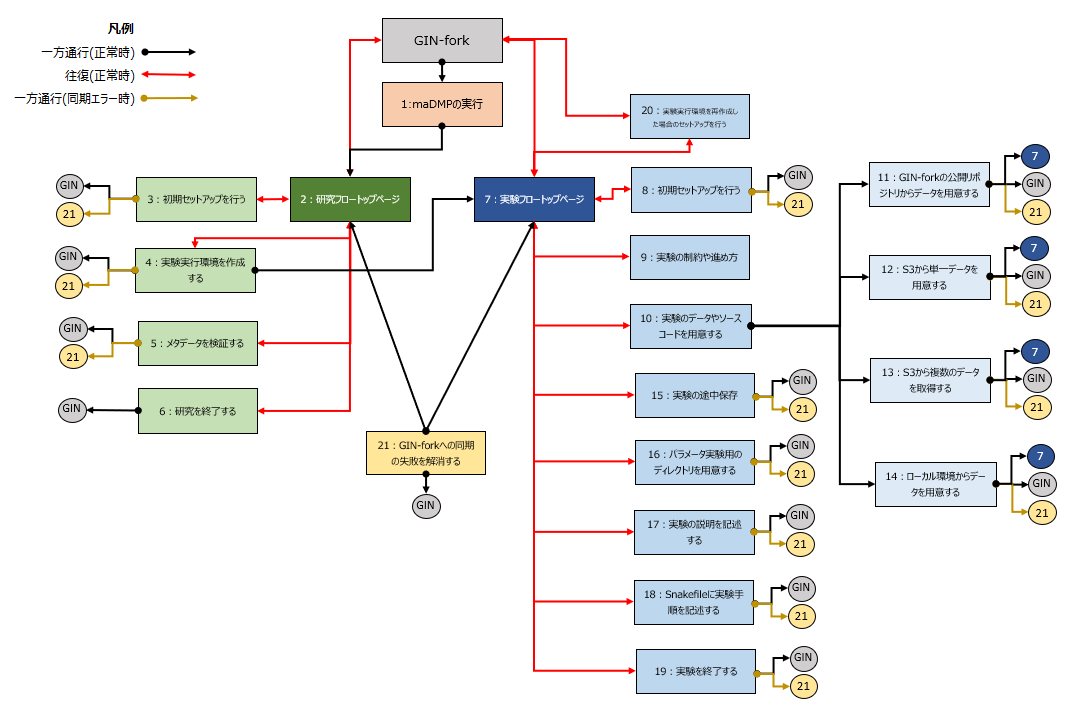
遷移イメージ
GIN-fork、タスクNotebookの遷移イメージは以下となります。
用語説明
データガバナンス機能で使用されている各用語について、イメージを用いて以下に記載します。
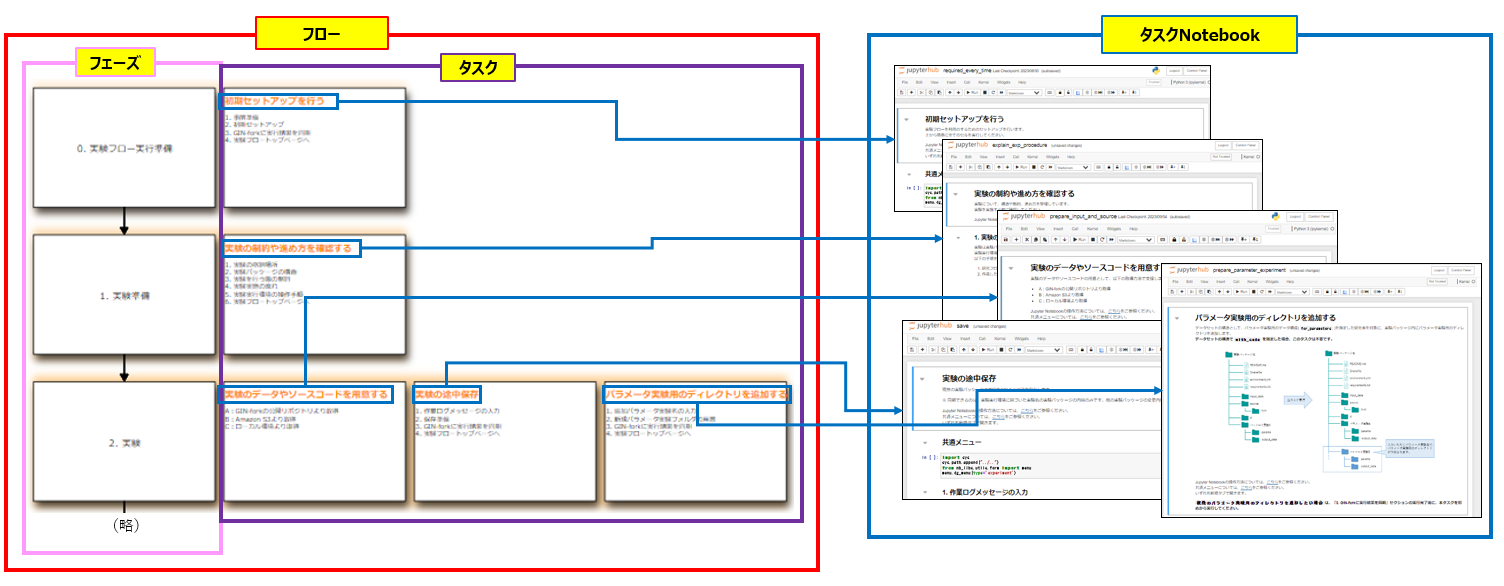
- フロー、フェーズ、タスク、タスクNotebook
- 折り畳み
複数のセルが折り畳まれ、ひとつにまとめられている状態を指します。

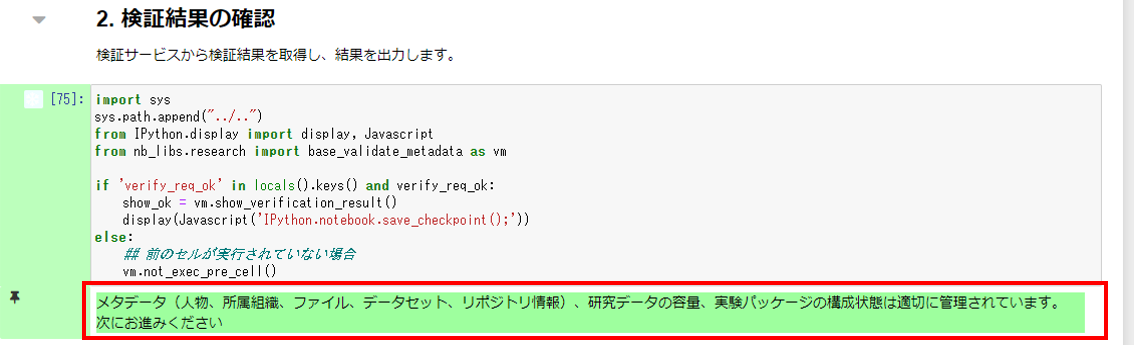
- ガイドメッセージ
セル実行後に出力されるメッセージを指します。

 ENGLISH
ENGLISH
